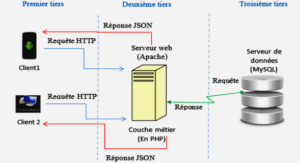
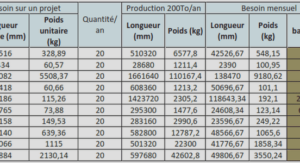
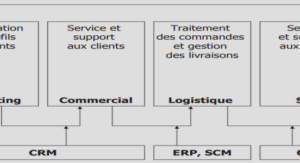
Comparaison de paires par alignement de séquences
Insérer ou supprimer du code entre la version originale et la copie d’un morceau de code source induit un impact sur la représentation de celui-ci, que ce soit sous une forme lexemisée ou par un arbre de syntaxe. Ainsi la recherche de facteurs exacts ou sous-arbres exacts, que nous allons aborder ultérieurement, se révèle inadéquate car découpant des correspondances pour cause de fossés non correspondants. Nous rappelons ainsi dans ce chapitre des techniques d’alignement de chaînes et d’arbres par programmation dynamique permettant de récupérer de telles correspondances approchées. Si celles-ci sont de complexité générale prohibitive pour la comparaison globale de projets, elles peuvent s’avérer intéressantes afin de raffiner la nature de zones de forte similarité pressenties par d’autres méthodes de filtrage de complexité temporelle plus avantageuse. Présentation La recherche de similarité entre deux projets exprimés sous la forme de chaînes de lexèmes peut bénéficier de méthodes d’alignement de séquences. Ces méthodes utilisant des techniques de programmation dynamique sont couramment utilisées en bioinfor- matique afin de déterminer des zones de similarité entre séquences biologiques (nucléotides d’ADN, ARN ou acides aminés) et quantifier leur niveau de différence ou de ressemblance afin de déterminer des phylogénies. Il s’agit ainsi schématiquement de déterminer les chaînes ajou- tées, supprimées ou modifiées entre les deux séquences comparées. Dans cet objectif soit une métrique de similarité, soit une métrique de distance entre deux séquences t), dans le second cas il s’agit du contraire. On note que le jeu d’opérations élémentaires présenté n’est pas minimal (dans la mesure où une substitution peut être remplacée par une suppression suivie d’un ajout) ; il pourrait également être étendu avec de nouvelles opérations si cela présente un intérêt pratique (substitutions d’une chaîne de longueur non-unitaire d’éléments, coûts personnalisés utilisant des matrices de coût…).
L’objectif d’une méthode d’alignement de lexèmes ne consiste pas à déterminer la LCS de deux unités lexemisées mais plutôt un ensemble de couples de facteurs exactement ou approximativement correspondants. Un couple de facteurs exactement correspondants présente une égalité lexème par lexème contrairement aux couples approxi- mativement correspondants possédant une distance d’édition non nulle. Le seul critère d’une distance d’édition seuil apparaît néanmoins limitatif pour juger de la pertinence d’un couple de facteurs correspondants. Celle-ci doit être mise en relation avec la longueur (voire le volume) des facteurs. La présence d’une zone de non-correspondance (fossé) importante peut égale- ment motiver la réductibilité d’un couple de correspondances, i.e. son découpage en plusieurs couples de plus petites longueurs n’intégrant pas le fossé.Il s’agit ensuite de sélectionner l’opération d’édition optimisant s à partir d’une des trois paires de sous-chaînes de u et v. Le coût de cette opération est alors ajouté pour s et celle-ci est ajou- tée à la séquence d’opération e. Nous constatons que le calcul récursif des valeurs de s et e pour les trois paires de sous-chaînes occasionne des calculs redondants avec jusqu’à 3 ) : la matrice est alors calculée par balayage par ligne, colonne, diagonale ou anti-diagonale. Fina- lement la séquence d’opérations d’édition optimale peut être obtenue par la détermination du chemin de la matrice partant de la cellule (m, n) (chaînes (u, v)) jusqu’à la cellule (0, 0) (chaînes (ǫ, ǫ)) en déduisant pour chaque cellule (i, j) l’opération d’édition optimale ainsi que la cellule prédécesseur parmi les trois cellules (i − 1, j), (i, j − 1) et (i − 1, j − 1). Le chemin représentant la séquence d’opérations sur la matrice n’est pas nécessairement unique.
Nous constatons ainsi que la détermination de la métrique de similarité s(u, v) nécessite mn opérations pour le remplissage de la matrice avec la nécessité de conserver en mémoire uniquement une ligne (calcul par balayage par ligne) ou une colonne (calcul par balayage par colonne) de la matrice et la cellule précédemment calculée. La reconstitution de la séquence d’opérations élémentaires d’édition optimale nécessite en revanche la conservation complète de la matrice, soit une mémoire de mn logOn notera que lorsque le calcul de la matrice est réalisé par balayage par anti-diagonale, seules les deux précédentes anti-diagonales sont nécessaires au calcul des valeurs de l’anti- diagonale courante : l’absence de dépendance à une cellule de l’anti-diagonale courante autorise le calcul simultané en parallèle de toutes les valeurs des cellules de cette anti-diagonale ce qui est un moyen avantageux d’exploiter certaines capacités d’unités de traitement graphiques (GPU) [46, 73], dans la limite de la taille de leurs tampons de calcul.