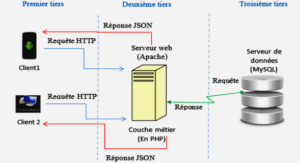
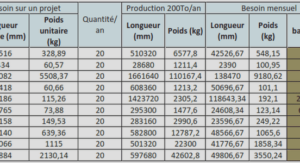
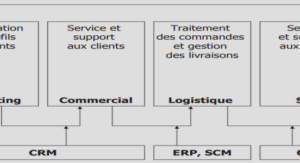
Impact des variations de la composition et de la structure du microbiote du sol
La plante et son microbiote racinaire forment un écosystème complexe dans lesquel plusieurs organismes contribuent au fonctionnement général et à la stabilité de l’écosystème (Hacquard & Schadt, 2015). La composition et la structure du microbiote racinaire sont très dynamiques et sont influencées par la disponibilité en nutriments, les paramètres environnementaux et les interactions d’origine biotiques (Llado et al., 2017 ; Haas et al., 2018). Si les paramètres édaphiques et l’activité de la plante hôte sont connus pour être fortement impliqués dans la structure du microbiote racinaire, les avis restent partagés concernant l’importance relative de ces deux facteurs dans la composition du microbiote racinaire (Lareen et al., 2016). D’une part, les facteurs dépendants de la plante hôte tels que les exsudats racinaires et les métabolites secondaires affectent la colonisation des racines par les micro-organismes. Ces facteurs peuvent dépendre du génotype de l’hôte mais aussi de facteurs extrinsèques tels que l’état de la plante et du sol ou encore les variations environnementales. D’autre part, les facteurs environnementaux tels que les propriétés du sol et le climat sont connus pour avoir un impact sur les communautés de micro-organismes du sol, principal réservoir de micro-organismes pour les racines (Fierer, 2017). Il a été démontré que le microbiote racinaire de la plante évolue rapidement en réponse aux variations abiotiques qui correspondent à des stress responsables de la réduction de la croissance de la plante (Lau & Lennon, 2012). De telles capacités sont très intéressantes dans le domaine de l’ingénierie microbienne pour optimiser la croissance des plantes tout en réduisant les effets néfastes des stress liés au changement climatique (Farrar et al., 2014; Meena et al., 2017). Pour ce faire, il est nécessaire d’identifier comment manipuler et contrôler le microbiote du sol, de comprendre comment la composition et les fonctions du microbiote racinaire sont régulées et, enfin, de comprendre quel est l’impact de la composition de ce microbiote racinaire sur la biologie de l’hôte. Si ces questions font aujourd’hui l’objet de travaux de recherches sur les plantes annuelles d’intérêts agronomiques (Chaparro et al., 2012), peu d’études concernent les arbres malgré un cycle de vie très différents et des capacités d’interactions plus larges par rapport aux plantes herbacées et annuelles (Nehls, 2008; Uroz et al., 2016; Liao et al., 2019). Une importante variabilité existe entre les capacités fonctionnelles des micro-organismes avec des redondances, des synergies et des antagonismes (Chaparro et al., 2012). Bien que les paramètres influençant la composition taxonomique du microbiote du sol et, dans une moindre mesure, ceux influençant la composition taxonomique du microbiote racinaire sont aujourd’hui bien connus, peu d’informations sont disponibles concernant l’ampleur de la redondance fonctionnelle de ces variations.
Dans ce contexte, les objectifs de ce chapitre sont, d’une part, de caractériser la composition et la structure du microbiote d’un sol de peupleraie durant deux années successives, et, d’autre part, d’évaluer si les potentielles modifications de la composition du microbiote du sol sont également visibles au niveau du microbiote et du métabolome racinaire du peuplier. Plusieurs questions se posent alors : Afin de répondre à ces questionnements, le sol issu d’une peupleraie âgée de dix-huit ans et dominée par Populus trichocarpa x deltoides a été prélevé durant deux années consécutives. Plus précisément, le sol a été collecté sous le même arbre en juin 2016 (Sol 1) et en juillet 2017 (Sol 2) afin de caractériser la composition et la structure des communautés bactériennes et fongiques du sol d’une année à l’autre. Ensuite, en cultivant des boutures de peuplier dans le Sol 1 et le Sol 2 en serre, nous avons caractérisé et comparé la composition et la structure des communautés microbiennes associées aux racines ainsi que le métabolome primaire et secondaire du Peuplier gris (Populus tremula x alba) successivement les deux années. Les communautés bactériennes et fongiques du sol et des racines ont été caractérisées par séquençage Illumina MiSeq haut débit des amplicons d’ADNr ITS et d’ARNr 16S tandis que le métabolome racinaire a été analysé par Chromatographie en phase Gazeuse couplée à la Spectrométrie de Masse (en anglais, Gas Chromatography- Mass Spectrometry ou GC-MS). Les résultats de cette étude sont décrits sous la forme d’un article scientifique publié dans la revue Phytobiomes en mars 2020. Les tableaux supplémentaires sont disponibles en Annexe (Annexe 3 de la page 27 à la page 52).