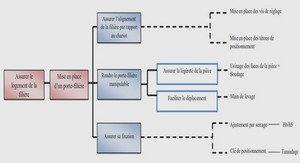
de de la flore bactérienne par la technique DGGE (Denaturing Gel Gradient Electrophoresis)
La seconde approche employée afin d’étudier la flore bactérienne présente dans les enrichissements sur ETBE et sur MTBE est la technique DGGE qui présente l’avantage de permettre l’identification des micro-organismes pour lesquels une bande est obtenue après électrophorèse puis découpage, amplification PCR, purification et séquençage. Après extraction de l’ADN des enrichissements et amplifications PCR, réalisées sur la région hypervariable V3-V4 des gènes codant l’ARNr 16S, les échantillons ont été déposés sur gel d’acrylamide, avec un gradient dénaturant d’urée/formamide 20-70 %. Le résultat de cette migration est présenté sur la figure 4.10. Chaque bande bien identifiable obtenue sur le gel a ensuite été découpée, purifiée puis amplifiée par PCR avec le même couple d’amorces (sans les GC-clamp), afin d’être séquencée. Les séquences des gènes codant l’ARNr 16S obtenues (400-500 bp) ont été comparées à celles de la base de données du site RDP afin d’identifier les micro-organismes correspondants. De même, des alignements de ces séquences ont été effectués en utilisant l’outil en ligne du site Greengenes. Ces alignements ont enfin été analysés avec le logiciel Seaview afin de réaliser des arbres phylogénétiques regroupant l’ensemble des micro- organismes présents dans les enrichissements sur ETBE ou dans les enrichissements sur MTBE. Ces arbres sont présentés en annexes sur les figures A.1 et A.2, respectivement. Les résultats de l’analyse DGGE sont discutés ci-dessous au cas par cas. a) Analyse comparative des enrichissements BE1-ETBE et BE1-MTBE
L’étude des capacités de dégradation a montré que pour BE1, celles-ci étaient différentes selon le substrat d’enrichissement. D’un côté, le toluène n’est pas dégradé par BE1-ETBE alors qu’il l’est par BE1-MTBE, et de l’autre côté, BE1-ETBE a démontré sa capacité à dégrader le m-xylène et le p-xylène, contrairement à BE1-MTBE. Ces différences sont le reflet des différences de composition phylogénétique de ces enrichissements. En effet, la comparaison des profils de migration DGGE entre les deux enrichissements montre une composition bactérienne différente malgré une origine commune. Huit bandes sont visibles dans BE1-ETBE, tandis que onze sont visibles dans BE1-MTBE. L’analyse des séquences et de leurs assignations montre également que parmi ces bandes, aucune n’est commune aux deux enrichissements, bien que les assignations correspondent aux mêmes phyla (Bacteroidetes, Alpha-, Beta-, Gamma- Proteobacteria, et Actinobacteria). Une souche en particulier, présente uniquement dans BE1-ETBE (bande 1g), correspond à Rhodococcus. Or, les souches Rhodococcus sp. IFP 2040 et IFP 2043, dégradant toutes deux l’ETBE, ont été isolées de BE1-ETBE. Des souches du même genre bactérien ont été étudiées pour leurs capacités au cours de différentes études passées (Auffret et al., 2009 ; Hernandez-Perez et al., 2001 ; Malandain et al., 2010), et la souche Rhodococcus wratislaviensis IFP 2016, pouvant dégrader l’ETBE et le MTBE, a été montrée responsable de la dégradation de plusieurs composés, notamment les BTEXs et des n-alcanes (Auffret et al., 2009). Il est donc probable que ces souches jouent un rôle important dans les capacités de dégradation de l’enrichissement BE1-ETBE.
Les résultats précédents ont montré que GE1-MTBE avait un spectre de dégradation pour les différents substrats testés plus important, notamment concernant la dégradation du toluène, et du m- et p-xylène, qui ne sont pas dégradés par GE1-ETBE, dégradant uniquement le benzène. Les capacités concernant les n-alcanes sont elles aussi différentes dans les deux cas. Ces différences sont là aussi le reflet d’une composition phylogénétique différente. En effet, la comparaison des profils de migration DGGE entre ces enrichissements montre une composition bactérienne différente malgré une origine commune. Seules quatre bandes ont été obtenues en DGGE pour GE1-ETBE, tandis que GE1-MTBE en présente huit. Néanmoins, deux bandes sont communes aux deux enrichissements: les bandes 2c et 6g, dont les séquences d’ADN partagent 84 % d’identité (2c: Gammaproteobacteria avec 100 % d’identité, et 6g: Proteobacteria avec 91 % d’identité), et les bandes 2d et 6i, dont les séquences d’ADN partagent 99 % d’identité (Piscinibacter sp., avec respectivement 99 % et 100 % d’identité). En outre, seules des bandes correspondant à des Proteobacteria sont visibles sur GE1-ETBE, tandis qu’une diversité plus importante est observée dans GE1-MTBE. Il faut noter que la souche Betaproteobacteria IFP 2047, capable de dégrader entièrement l’ETBE, et isolée à partir de GE1-ETBE, correspond bien à la bande 2b (99 % d’identité au niveau des séquences des gènes codant l’ARNr 16S). Il est donc probable que cette souche joue un rôle important dans la dégradation de ce substrat dans cet enrichissement. Nous ne retrouvons pas de bande correspondant à Rhodococcus alors que nous avons isolé Rhodococcus sp. IFP 2041 de GE1-ETBE. Il est possible qu’elle soit minoritaire dans la population. Concernant GE1-MTBE, la séquence de la souche de Proteobacteria IFP 2052 comporte 96 % d’identité avec la séquence de la bande 6i (Piscinibacter), il est donc probable que cette souche joue bien un rôle dans la dégradation du MTBE dans cet enrichissement. Néanmoins, le problème de sa caractérisation reste posé au vu des résultats présentés précédemment (RDP: Gammaproteobacteria ; NCBI Beta- et/ou Gamma- Proteobacteria). Le phylum Gammaproteobacteria est par ailleurs largement représenté, avec 4 bandes. Plusieurs souches appartenant à ce phylum ont été décrites dans la littérature pour leur capacité à dégrader les BTEXs, notamment Pseudomonas putida (Mazzeo et al., 2010) ou Pseudoxanthomonas spadix BD-a59 (Lee et al., 2012). La présence de micro-organismes appartenant à ce même phylum dans cet enrichissement pourrait expliquer les capacités de dégradation accrues pour les BTEXs par rapport à son homologue enrichi sur ETBE.