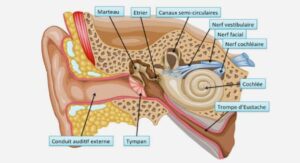
Étude et suivi de la résistance des virus influenzae A aux inhibiteurs de la neuraminidase
Production et culture des baculovirus
Un système de transfection de cellules Sf9 à partir d’un bacmid a été utilisé pour générer des baculovirus contenant nos neuraminidases d’intérêts. Les cellules Sf9 ont été transfectées par 2 μg de bacmid en présence de Cellfectin (Système Bac-to-Bac®) dans un milieu Grace’s insect medium (Gibco, life technologies) sans aucune supplémentation. Vingt-quatre heures après la transfection, le milieu a été changé par du milieu de croissance composé de Sf900 II supplémenté avec 10% de SVFi, 1% PS et 0,1% de pluronic F68 (Sigma-Aldrich). Les surnageants contenant le baculovirus ont été récoltés 96 heures après la transfection et séquencés (Voir paragraphes 3.2.11., ci-dessous) (Figure 22). 186 Le surnageant issu de la transfection (P0) (1 mL) a été utilisé pour infecter 10,0×106 de cellules Sf9 (flasque de 175 cm2 ). La récolte du P1 a eu lieu à 72 heures post-infection en fonction de l’aspect des cellules. Après titrage du baculovirus, le surnageant P1 a été utilisé pour infecter 1,0×109 de cellules Sf9 (MOI=0,1) dans 1 L (réparti entre 4 erlens de 1 L) de milieu d’infection. Le surnageant P2 a ensuite été concentré par une ultracentrifugation à 160 000 g pendant deux heures et demie à 4°C dans une ultracentrifugeuse optima L-80 XP (Figure 22). Le baculovirus a été repris en PBS 1X avant d’être titré, séquencé et stocké à 4°C (Voir paragraphes 3.2.7. et 3.2.11., ci-dessous). Figure 22 : Représentation schématique des étapes de production et de purification des neuraminidases recombinantes.
Titrage en plage de lyse du baculovirus
Des cellules Sf9 ont été ensemencées à raison de 1,0×106 cellules (environ 50% de confluence) par puits dans des plaques six puits. Ces cellules ont ensuite été infectées par des dilutions en séries de raison dix de surnageant contenant le baculovirus. Après une incubation de deux heures sous agitation douce, les inoculas ont été retirés et les cellules recouvertes par un mélange de milieu Sf900 II 1,3X (Gibco, life technologies) et d’agarose 4% (Gibco, life technologies). Une fois solidifié, 2 mL de milieu Sf900 II supplémenté avec 10% SFVi a été ajouté dans chaque puits. Les plaques ont été visualisées 96 heures après une incubation à 27°C sans CO2 (Figure 22). Les plages de lyse ont ensuite été dénombrées après coloration par une solution de rouge neutre à 0,1 mg/mL. Les titres ont été calculés avec la formule suivante : ൈ݊ ݅ݐݑ݈݅݀݁݀ݎݑ݁ݐ݂ܿܽ ൈ݁ ݏݕ݈݈݁݀݁݃ܽ݁݀݁ݎܾ݉݊ ൌ ሻ Τ ݈݉ ݑ݂ሺ݁ݎݐ݅ݐ ͳ ᇱ݉݁݀ݑ݈ܸ ݏݐ݅ݑݎܽ݉ݑ݈ݑܿ݊݅ 3.2.8. Production et purification des protéines recombinantes Les neuraminidases recombinantes ont été produites après infection de cellules High Five à forte MOI. Succinctement, le baculovirus P2 ultracentrifugé a été utilisé pour infecter 2,0×109 de cellules High Five (MOI=5) dans 1 L (répartis en 4 erlens de 1 L) de milieu Express five supplémenté par de la L-Glu (20 mM). Après une incubation de 50 heures à 27°C sans CO2 avec une agitation douce, la récolte a été centrifugée pendant 20 minutes à 5 500 g pour éliminer les débris cellulaires. Les surnageants ont ensuite été filtrés à 0,22 μm avant d’être concentrés par filtration tangentielle sur une cassette vivaflow 200 (seuil à 10 kDa) (Sartorius). Le concentrat a été purifié par incubation avec 4 mL de résine Nickel (Qiagen) permettant la récupération des protéines taguées avec de l’histidine lors d’une élution avec de l’imidazole (500 mM, Sigma-Aldrich). Les 14 mL d’élution ont ensuite été concentrés et lavés par centrifugation dans des amicons® ultra 15 (seuil à 30 kDa, Millipore). Après dosage des protéines totales (Bradford, vWR, life science), les 500 μL de protéines concentrées ont été incubés à température ambiante sur la nuit avec de la thrombine (1 U par mg de protéine, Thrombine cleavage capture Kit, Novagen). Finalement, après élimination de la thrombine par affinité avec des billes de streptavidine, les neuraminidases ont été analysées et purifiées par chromatographie d’exclusion de taille sur une colonne SuperdexTM 200 Increase 10/300GL (GE). Les fractions protéiques correspondant aux tétramères de neuraminidase ont été récupérées. L’identification et la pureté des protéines ont été évaluées avant (gel natif, Mini protean TGX gradient 4 à 20%, Biorad) et après dénaturation (gel, Mini protean TGX 12%, Biorad) par une coloration au bleu de Coomassie et par la réalisation de western blots avec des anticorps anti-histidine (Cusabio) et des anticorps anti-N9 (BEI ressources). Enfin, la quantification des protéines a été effectuée par la méthode de Bradford et la caractérisation des propriétés enzymatiques a été réalisée par des techniques fluorimétriques (Voir paragraphe 3.2.9. et 3.2.10., ci-dessous). 188 3.2.9. Mesure de l’activité sialidasique et test d’inhibition enzymatique L’oseltamivir, le zanamivir et le laninamivir ont respectivement été fournis par Roche, Ltd., GlaxoSmithKline et Daiichi-Sankyo. Les tests d’activité sialidasique et d’inhibition enzymatique ont été réalisés à l’aide d’un fluorimètre FLUOstar OPTIMA (BMG LABTECH) comme décrit dans une étude précédente (272). Les activités sialidasiques totales ont été calculées comme la quantité de substrat, l’acide 2′-(4-méthylumbelliféryl)-α-DN-acétylneuraminique (MUNANA) (Sigma), dégradée en 4-méthylumbelliférone (4-Mu) en une heure par mL de suspension virale (4-Mu-nmol/h/mL). Le test d’inhibition enzymatique de la neuraminidase a été réalisé en utilisant une quantité d’activité totale normalisée (10 4-Mu-nmol/h/mL). Les CI50 ont été calculées à l’aide du logiciel GraphPad (Prism). L’interprétation de l’inhibition enzymatique des neuraminidases par les INA est basée sur l’augmentation de la CI50 par rapport à celle des virus sensibles (Voir Partie A, Chapitre 3). 3.2.10. Mesure des cinétiques enzymatiques (Km) Le Km a été évalué en utilisant le substrat MUNANA comme décrit précédemment (6,273). La fluorescence du 4-Mu a été mesurée chaque minute pendant une heure avec un fluorimètre FLUOstar Optima (BMG Labtech). La vitesse initiale a été calculée pour chaque concentration de substrat et intégrée dans une équation non linéaire de Michaelis-Menten par le programme MARS (BMG) pour calculer le Km.
Séquençage
L’extraction de l’ADN du baculovirus, à partir des surnageants du premier et dernier passage sur cellules Sf9, a été réalisée à l’aide du kit Gentra® Puregene® Blood Core Kit (Qiagen). Une PCR ciblant la région insérée dans le baculovirus ou dans les bacmids a été réalisée à l’aide de l’ADN polymérase Q5® High Fidelity Hot start et d’amorces spécifiques (Voir Annexe 1, SV40 polyA reverse et Polyhedrin forward). Le séquençage par Sanger de l’amplicon ainsi que de tous les Bacmids a été réalisé sur des analyseurs ABI Prism 3730XL (Genoscreen Lille, France). Les séquences ont ensuite été analysées à l’aide du logiciel MEGA7 (274). Pour les virus recombinants porteurs d’une N9, après extraction sur plateforme automatisée EasyMag (bioMérieux), chaque segment génomique a été rétro-transcrit et amplifié en utilisant un protocole de RT-PCR multiplex préalablement publié (275). La bibliothèque d’ADN a été préparée à l’aide du kit Nextera™ XT (Illumina) consistant en une fragmentation enzymatique, à l’ajout de codes-barres par PCR (12 cycles) et à une sélection de taille des fragments par des billes magnétiques. Les segments ont ensuite été séquencés à l’aide d’une plateforme de séquençage Illumina NextSeq 500® (Illumina) par une cartouche MidOutput (Illumina). L’analyse des résultats de séquençage a été réalisée comme décrite précédemment en utilisant un pipeline informatique standardisé (276,277).
Analyses statistiques
Les résultats des activités sialidasiques, des Km et les mesures de CI50 ont été analysés par un test de Mann–Whitney en utilisant le logiciel GraphPad (Prism). Les résultats ont été considérés comme significativement différents si p ≤ 0,05. 3.3. Optimisation de la production de neuraminidases recombinantes Au cours de cette thèse, le travail de mise au point s’est principalement concentré sur la production des neuraminidases ; du montage génétique à la purification et l’analyse de ces protéines recombinantes. La production des bacmids d’intérêts avait déjà été mise au point par le Dr Émilie Frobert ne nécessitant pas d’adaptation pour notre application à l’exception d’un changement de kit d’extraction. En effet les bacmids et baculovirus (bMON14272 du système Bac-to-Bac®) ayant un génome à ADN de taille supérieure à 135 kilobases (kb) nous avons dû utiliser les kits Gentra® Puregene® Blood Core Kit (Qiagen) ou NucleoBond® Xtra Midi (Macherey-Nagel) permettant l’extraction d’ADN de taille allant jusqu’à 300 kb alors que nous utilisions des kits ayant une limite d’extraction à environ 50 kb. A l’inverse, l’obtention d’une concentration de baculovirus suffisante pour permettre une infection des cellules High Five dans des conditions optimales et la production de protéines recombinantes en quantité suffisante a nécessité un grand nombre d’adaptations des paramètres d’infections et de culture de ces virus. Nos premiers essais ont été réalisés avec des cellules d’insectes Sf9 et High Five adhérentes et entretenues en flasques. La difficulté de maintien de ces lignées ainsi que l’impossibilité d’augmenter leur production, nous ont obligés à rapidement envisager une culture en suspension de ces cellules. Suite à la transfection du bacmid dans les cellules Sf9, nous avons testé plusieurs temps de récolte à 72, 96 heures et sept jours. Les titres viraux obtenus étant croissants et dépendants du délai de récolte, nous avons choisi la condition intermédiaire de récolte à 96 heures permettant d’allier un temps d’incubation court et une bonne production des baculovirus (Figure 23A). Lors du premier passage (P1) d’amplification des baculovirus, le temps d’incubation était adapté à la lyse cellulaire observable et nous avons testé une infection soit par une MOI standardisée à 0,1 soit par un volume de 1 mL de surnageant issu de la transfection (P0). Les résultats étant similaires nous avons choisi de faire notre première amplification en utilisant directement un volume de 1 mL de surnageant, économisant ainsi le temps de titrage du P0 (Figure 23B). Après ce P1 qui permet une augmentation des volumes de surnageant contenant le baculovirus, nous avons réalisé un deuxième passage d’amplification (P2) dans le but d’augmenter notre titre viral pour obtenir un titre supérieur à neuf log10pfu/mL. Nous avons dans un premier temps testé différents temps de récolte (3, 4, 5, 6 et 7 jours) mais en l’absence de différence sur le titre viral nous avons choisi de récolter dès 72 heures et d’ajouter une étape de concentration du virus par ultracentrifugation (Figure 23C). Une fois les baculovirus concentrés obtenus, des tests de productions de protéines recombinantes sur cellules High Five ont pu être réalisés. Cette production doit se faire après une infection à forte MOI, nous avons donc testé les MOI de 5 et 10 pour finalement sélectionner une MOI de 5 afin de limiter les volumes de baculovirus utilisés à chaque production. La densité des cellules a également été testée et malgré une forte variabilité sur la quantité de protéines produites d’une expérimentation à l’autre, nous avons pu sélectionner la densité de 2,0×106 cellules High Five par mL comme étant celle avec le meilleur rendement (Figure 23D). Enfin, la réalisation de cinétique de production de la neuraminidase recombinante a permis de fixer un temps de récolte à environ 50 heures pour obtenir l’activité totale maximale mais également la meilleure activité normalisée à la concentration de protéines totales du milieu permettant de limiter les contaminations par les protéines cellulaires et donc d’être dans des conditions favorables lors de la purification (Figure 23E et F)
Virus réassortants
Après la double concentration, les virus recombinants porteurs soit de la N9 WT, soit de la N9 substituée en R292K ont été obtenus respectivement à des titres infectieux de 8,65 et 7,78 log10TCID50/mL et à des titres hémagglutinants de 1 458 et 13 122 UHA (Tableau 3). Les activités enzymatiques ont été normalisées par rapport au titre infectieux et sont exprimées en 4-Mu-nmol/h/log10TCID50. La différence entre les activités des virus n’est pas significative en raison de l’utilisation d’un test non paramétrique (Mann–Whitney) (tripliquât par virus). Néanmoins, l’activité des virus porteurs de la N9 WT (198,0 ± 17,5 4-Mu-nmol/h/log10TCID50) est six fois plus importante que celle des virus porteurs de la N9 substituée (36.2 ± 17,5 4-Mu-nmol/h/log10TCID50) (Figure 27A). Le Km représente l’affinité de la neuraminidase pour son substrat, le MUNANA. Pour interpréter les Km, il ne faut pas oublier que plus le Km est élevé plus l’affinité est basse et réciproquement. La présence de la substitution R292K dans la N9 s’accompagne d’une chute significative de l’affinité Les CI50 ont été interprétées selon les recommandations de l’OMS en fonction du rapport entre la valeur des CI50 du virus substitué et la valeur des CI50 du virus sensible. La substitution R292K entraine une augmentation d’au moins 14 000 fois la valeur des CI50 de l’oseltamivir pour les virus porteurs d’une N9 classant ces virus comme ayant une sensibilité très diminuée. L’impact de cette substitution sur la résistance au zanamivir et au laninamivir semble plus modéré mais les virus restent classés comme ayant une sensibilité diminuée à ces molécules avec des ratios de 37 pour le zanamivir et de 20 pour le laninamivir (Tableau 3, Figure 28
Introduction et objectifs |