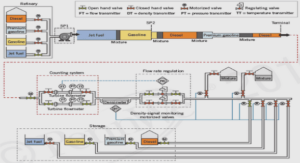
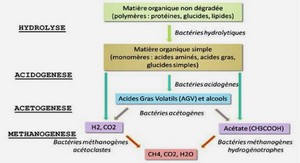
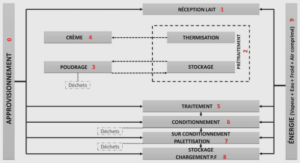
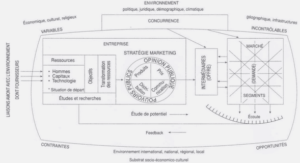
Bagage de phylogénie moléculaire
La phylogénie est une discipline scientifique qui étudie les « parentés entre différents êtres vivants en vue de comprendre l’évolution des organismes vivants » 1 . Les premières phylogénies (Charles DARWIN, 1809-1882 ; Ernest HAECKEL, 1834-1919) se basaient sur des caractères morphologiques, anatomiques et/ou physiologiques afin de comparer les organismes vivants et d’étudier leur parenté. Mais lorsqu’il s’agit de comparer des organismes bactériens ou viraux ces critères de comparaison atteignent leur limite. Depuis le développement de la biologie moléculaire et la découverte de l’ADN (acide désoxyribonucléique) comme support de l’hérédité dans les années cinquante, de nouveaux caractères sont utilisés comme source d’information pour l’inférence de phylogénies : les séquences de macromolécules (ADN, ARN et protéines). Les premières études phylogénétiques essentiellement basées sur des séquences protéiques remontent au début des années soixante et donnent ainsi naissance à une nouvelle branche de la phylogénie : la phylogénie moléculaire. Mais ce n’est que vers la fin des années soixante-dix, avec le développement de techniques spécifiques permettant de séquencer des fragments d’ADN à grande échelle et à faible coût que la phylogénie moléculaire connait un essor grandissant. En particulier parce que cette discipline est très utilisée en génomique fonctionnelle, science qui étudie le rôle des gènes. La phylogénie moléculaire est aussi très utilisée par les épidémiologistes car elle permet de mettre en évidence des liens entre différentes souches virales, liens qui reflètent des chaînes de transmission. Un exemple souvent cité car c’est le premier qui utilise des outils de phylogénie moléculaire dans un cadre médico-légal, est celui d’un dentiste de Floride, séropositif, qui est suspecté être la source de contamination de quelques uns de ses patients (Ou et al, 1992). Les indices ayant menés à cette hypothèse proviennent d’une patiente atteinte du syndrome de l’immunodéficience acquise (SIDA) mais pour laquelle aucune situation de contamination n’a pu clairement être identifiée, hormis deux interventions chirurgicales venant de son dentiste. Pour confirmer un éventuel lien épidémiologique, des souches virales ont été prélevées chez le dentiste, chez la patiente, ainsi que chez six autres patients qui ont séroconverti pendant l’enquête ; par ailleurs, trente-cinq souches virales provenant d’individus locaux ont été rajoutées comme souches témoins. L’analyse phylogénétique de toutes ces souches virales a révélé que la souche collectée chez le dentiste est phylogénétiquement très proche de celles collectées chez ses patients, confirmant ainsi la source de contamination. Mais le mode de contamination reste indéterminé. De nombreux autres exemples comme celuilà sont disponibles dans la littérature, Leitner et Fitch (1999) en commentent d’autres.Dans ce chapitre, nous présentons brièvement les différentes méthodes d’inférence phylogénétique. Mais avant cela, nous présentons les bases de données biologiques, véritables sources d’information pour les études moléculaires, puis l’étape d’alignement, fondamentale à toute analyse phylogénétique. Enfin, nous terminerons ce chapitre par l’exposé de quelques méthodes de parcimonie permettant de reconstruire les annotations ancestrales (par exemple des régions géographiques) à partir d’une phylogénie et des annotations associées aux feuilles de cette phylogénie qui représentent les souches virales de l’alignement. Des compléments d’information peuvent être trouvés dans les ouvrages de Lemey et al. (2009b) ou celui de Felsenstein (2003).
Bases de données biologiques
Les études de phylogénie moléculaire sont souvent basées sur des séquences nucléotidiques. Pour être facilement accessibles, et pour faciliter le traitement de l’information, les séquences nucléotidiques obtenues par les biologistes sont stockées dans des bases de données. Ces bases de données fournissent aussi une pléthore d’outils pour manipuler ou analyser les séquences, mais aussi des informations supplémentaires sur chacune d’elles. Ces informations, ou annotations, sont très utiles car elles renseignent sur l’organisme de collecte, les propriétés de la séquence, les auteurs, etc., permettant ainsi de cibler les recherches dans ces bases. Il existe de nombreuses bases de données biologiques mais la plupart sont spécifiques à un organisme, une fonction, etc. Toutefois, il existe trois bases de données principales : – EMBL-Bank (European Molecular Biology Laboratory), maintenue par EMBL-EBI (European Bioinformatics Institute) à Hinxton au Royaume-Uni ; – GenBank, maintenue par NCBI (National Center for Biotechnology Information) à Bethesda aux États-Unis ; – DDBJ (DNA Data Bank of Japan), maintenue par NIG/CIB (National Institute of Genetics, Center for Information Biology) à Mishima au Japon. Ces trois bases de données collaborent ensemble afin de partager les nouvelles soumissions ou les éventuelles mises à jour. L’ensemble des séquences nucléotidiques publiées y est donc accessible. Chaque séquence soumise se voit attribuer un numéro d’accession unique (qui reste le même quelle que soit la base de données) et qui permet de désigner, sans ambiguïté, les séquences dans la littérature. Par convention, les séquences nucléotidiques sont stockées sous le format de l’ADN, mais les bases de données contiennent aussi des séquences d’ARN (acide ribonucléique). Dans ce cas, ces dernières sont codées avec un « T », qui signifie la thymine, à la place d’un « U », pour designer l’uracile.Dans nos études, nous utilisons la base de données spécifique au VIH maintenue par le laboratoire national de Los Alamos : HIV Databases (www.hiv.lanl.gov). Elle met à disposition un grand nombre de séquences nucléotidiques du VIH de type 1 (VIH-1), du VIH de type 2 (VIH-2) et même du SIV (simian immunodeficency virus), virus analogue au VIH mais infectant naturellement les singes d’Afrique. Mise à jour périodiquement, elle contient toutes les séquences soumises dans GenBank, avec un décalage de quelques mois sur les dernières entrées de GenBank. En revanche, les séquences sont annotées avec plus d’informations que celles disponibles via GenBank, comme l’origine géographique de collecte, l’année d’isolation, le sous-type d’appartenance, le groupe à risque de l’individu chez lequel elle est prélevée, etc. Ces informations sont récupérées dans les publications correspondantes aux séquences par les gestionnaires de la base de données. De plus, le site internet propose une interface de recherche conviviale, ergonomique et adaptée aux particularités du VIH et du SIV. Il est ainsi très facile d’obtenir des séquences sur une région précise du génome, provenant d’un même pays ou d’un même continent, isolées chez un patient avec un facteur à risque particulier, etc. Des outils sont aussi mis à disposition et permettent le traitement spécifique de séquences du VIH/SIV, comme, par exemple, Sequence Locator qui permet de retrouver les coordonnées de début et de fin d’une séquence sur le génome de référence (HXB2 pour le VIH et SIVmm239 pour le SIV). Malgré le soin apporté au classement et au référencement des séquences, ces bases de données peuvent contenir des informations erronées. Il revient à l’utilisateur de vérifier la justesse des informations.
L’alignement, une étape indispensable
L’alignement de séquences nucléotidiques est une étape clef des études de phylogénie moléculaire. Cette étape ne peut se faire qu’avec des séquences homologues, c’est-à-dire des séquences nucléotidiques partageant un même ancêtre commun, puisqu’elle consiste à identifier, pour chaque séquence, les nucléotides dérivant du même nucléotide ancestral et à les positionner en regard. Le résultat de cette étape est l’obtention d’une matrice, appelée alignement, où chaque ligne correspond à une séquence et où chaque colonne, appelée site, contient les nucléotides dérivés d’un même nucléotide ancestral (Figure 1). Dans certaines séquences de l’alignement des gaps (ou indels) ont pu être introduits. Ils correspondent aux phénomènes biologiques d’insertions (ajout d’un ou de plusieurs nucléotides) ou de délétions (perte d’un ou de plusieurs nucléotides) qui se sont produits au cours de l’évolution. Toutefois, l’utilisation de gaps dans un alignement doit être faite avec parcimonie. Ainsi, un bon alignement est défini comme un alignement qui contient le moins d’évènements de mutation possibles,avec des pondérations différentes pour les différents évènements mutationnels (substitution, insertion, délétion, ouverture de gap, prolongation de gap, etc.). Comme l’alignement est la base de toutes méthodes de phylogénie moléculaire, il est indispensable d’avoir un alignement d’une qualité optimale afin d’inférer des phylogénies fiables. Dans le cas contraire, elles peuvent contenir des erreurs ou être aberrantes. C’est pour cela que les biologistes ôtent de l’alignement les sites les plus incertains, comme ceux contenant des gaps ou les parties trop divergentes (souvent en début ou en fin de l’alignement). Des méthodes automatisées existent pour résoudre des alignements. La plus simple concerne l’alignement entre deux séquences en se basant sur la distance d’édition (ou distance de Levenshtein). Cette distance mesure la similarité entre deux mots. Pour cela, elle calcule le nombre minimum de remplacements (ou substitutions), de délétions ou d’insertions nécessaires pour transformer un mot en l’autre. Rappelons que les séquences nucléotidiques peuvent être vues comme des mots sur l’alphabet génétique . Un algorithme simple de programmation quadratique permet de calculer la distance d’édition en , où et sont les longueurs respectives des deux séquences. Néanmoins cet algorithme calcule uniquement la distance (ou le score) de l’alignement optimal. Un algorithme supplémentaire est nécessaire afin d’en déduire l’alignement, il se fait en en réutilisant le tableau construit lors du calcul de la distance d’édition. Lorsque l’on souhaite aligner plus de deux séquences simultanément, le problème devient très vite complexe. Il est bien sûr possible d’adapter l’algorithme précédent dans le cas de plusieurs séquences, mais la complexité devient alors exponentielle sur le nombre de séquences, et l’application sur plus de quatre ou cinq séquences est inenvisageable. Pour contrer ce problème, des heuristiques sont proposées mais elles ne permettent pas de résoudre avec exactitude le problème de l’alignement. Les biologistes utilisent donc ces heuristiques afin d’obtenir une base convenable de l’alignement, puis le modifient manuellement avec des logiciels d’éditions. De nombreux programmes sont disponibles pour résoudre le problème d’alignement multiple de séquences. Une liste exhaustive est trouvée dans Lemey et al. (2009b). Dans nos études, seul le logiciel MAFFT (Katoh et al, 2005) est utilisé car il a été démontré qu’il est l’un des plus performants (Thompson et al, 2011).
Modèles d’évolution moléculaire
La distance évolutive entre deux séquences nucléotidiques est définie comme « le nombre moyen de substitutions par site s’étant produites depuis que ces séquences ont divergé de leur ancêtre commun » (Perrière & Brochier-Armanet, 2010). Pour calculer la distance évolutive qui sépare deux séquences dans l’alignement, une approche simpliste consisterait à compter le nombre de dissemblances (c’est-à-dire le nombre de sites différents) et de le diviser par la longueur de l’alignement. Cette distance évolutive est appelée -distance (exprimée en substitutions par site) et correspond à la distance observée entre les deux séquences, et non à la distance évolutive réelle. En effet, imaginons qu’entre deux séquences données, et sur un site donné, les nucléotides A et G sont observés. La -distance comptabilise une substitution, car c’est ce qui est observé. Mais si la base A est remplacée par la base T, puis par la base G, il y a eu deux évènements de substitutions réelles, mais toujours une substitution observée. Donc la -distance sous-estime la distance évolutive réelle, puisque de nombreuses substitutions cachées ont pu se produire. Des modèles d’évolution sont donc proposés pour estimer au mieux la distance évolutive réelle à partir de la distance évolutive observée. Ces modèles font les hypothèses simplificatrices suivantes : – les séquences évoluent uniquement avec un processus de substitution nucléotidique, c’est-àdire que les évènements d’insertion et de délétion ne sont pas pris en compte ; – les sites de l’alignement sont indépendants les uns des autres, c’est-à-dire que les évènements évolutifs d’un site n’ont aucune influence et ne sont pas influencés par les évènements évolutifs des autres sites de l’alignement ; – le processus d’évolution est markovien d’ordre 1, c’est-à-dire que l’état futur d’un site ne dépend que de son état actuel et non des états passés précédents ; – le processus d’évolution est identiquement distribué, c’est-à-dire qu’il est le même quel que soit le site de l’alignement ;