Exploration et analyse immersives de données moléculaires guidées par la tâche et la modélisation sémantique des contenus
Acteurs et processus de la biologie moléculaire
Les biomolécules sont impliquées dans le fonctionnement des organismes vivants et plus particulièrement de leur sous-unité la plus importante : la cellule. On retrouve parmi ces biomolécules, les molécules d’eau, qui constituent souvent la part majoritaire dans la composition des organismes, les lipides, qui sont les composants de base des membranes cellulaires permettant de créer des cloisons et compartiments, les acides nucléiques qui sont les constituants de l’ARN et de l’ADN, support de l’information génétique, les acides aminés qui forment les protéines, principaux acteurs du fonctionnement cellulaire, les sucres qui jouent un rôle fondamental dans de nombreux processus, puis diverses autres molécules, par exemple des cofacteurs comme l’hème. Nos travaux de recherche portent sur les biomolécules de plus grande taille, appelées également macromolécules, comme l’ADN et les protéines. L’information génétique stockée dans l’ADN est transmise et conservée de génération en génération grâce à la reproduction. Les protéines sont à la fois les ouvriers, les briques et les messagers impliqués dans le fonctionnement cellulaire. Nous porterons une attention particulière aux édifices macromoléculaires composés de plusieurs protéines appelés complexes protéiques, même si les résultats de nos travaux de recherches s’appliquent à une large gamme de biomolécules, des plus simples aux plus complexes.
Les biomolécules au coeur de la machinerie cellulaire
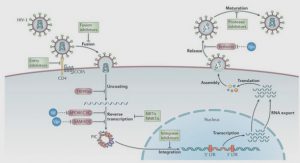
L’information génétique (génotype) est un plan dont l’exécution conditionne l’apparence d’un être vivant, son fonctionnement, et son comportement dans son environnement (phénotype). Cette information génétique est stockée de manière pérenne et reproductible sur un support de nature moléculaire : l’ADN pour Acide Desoxyribo-Nucléique ou l’ARN pour Acide Ribo-Nucléique. L’ADN est porté par les chromosomes, situés dans le noyau de la cellule (voir Figure 1.1). L’exécution de ce plan s’effectue à l’échelle moléculaire et commence par la lecture de l’information génétique dans le noyau et se termine par la production de toutes les protéines dans le cytoplasme (voir Figure 1.1). Ce processus de transformation de l’information génétique en des composants fonctionnels est commun à tous les êtres vivants. Le fonctionnement d’une cellule vivante implique aussi d’autres acteurs. Parmi ces molécules, les polysaccharides et les lipides ne sont pas générés par le code génétique, mais jouent un rôle prépondérant dans la structuration, notamment de la membrane cellulaire (voir Figure 1.1), d’autres stockent l’énergie nécessaire à la cellule et enfin certaines fonctionnent comme messagers inter- et intracellulaires
L’ADN
L’Acide Désoxyribo-Nucléique (ADN) est une biomolécule pouvant être considérée comme le plan de construction de tous les êtres vivants. Le support moléculaire contenant l’information génétique est une longue séquence de nucléotides, de quatre types : l’Adénine, la Thymine, la Guanine et la Cytosine. Ces nucléotides partagent une structure moléculaire commune constituée d’un sucre, le désoxyribose, et d’un groupe phosphate (voir Figure 1.2a). À cette partie commune se lie une base azotée spécifique à chacun des 4 types de nucléotides. Les nucléotides s’organisent en séquence de deux brins en complémentarité en établissant des liaisons hydrogènes spécifiques. L’Adénine et la Guanine se liant respectivement à la Thymine et la Cytosine. Les deux brins de l’ADN adoptent une structure hélicoïdale. Leur complémentarité permet tout d’abord d’assurer une certaine résistance de la structure à la dégradation et, en cas d’endommagement d’un des deux brins, la redondance de la complémentarité permet la réparation du brin intact. L’ADN est lui-même structuré de façon plus complexe, d’abord compacté par les histones (voir Figure 1.1), protéines structurantes possédant une forte affinité avec l’ADN qui s’enroule autour, puis finalement organisé en superstructures, les chromosomes. Chez les eucaryotes, organismes pluricellulaires possédant un noyau dans la cellule, l’ADN est stocké dans le noyau.
L’ARN
L’Acide RiboNucléique (ARN) est une biomolécule structurellement proche de l’ADN comportant néanmoins quelques différences. La première se retrouve au niveau de la séquence d’acides nucléiques qui, contrairement à l’ADN, très majoritairement composé d’un double brin sous forme d’hélice, s’organise en simple brin. Une seconde différence concerne les sucres constituant chacun des nucléotides puisque le désoxyribose de l’ADN est remplacé par un ribose pour l’ARN. La différence entre les deux groupements est illustrée dans la Figure 1.4. De plus, la Thymine présente dans l’ADN n’existe pas dans l’ARN et est remplacée par l’Uracile, complémentaire, comme la Thymine, à l’Adénine.
Les protéines
Les protéines sont les biomolécules considérées comme les acteurs moléculaires fonctionnels de la cellule. Les protéines sont à la fois les briques, les ouvriers et les messagers participant au fonctionnement cellulaire. Les règles qui régissent la production de protéines à partir de la lecture de l’information génétique sont décrites dans le code génétique universel, commun à tous les organismes vivants. Certaines assurent un rôle structurel, en étant notamment impliquées dans la construction et la structure du squelette de la cellule, comme l’actine et le collagène qui assurent le maintien physique et structurel de la cellule ainsi que la résistance de la matrice extracellulaire. Certaines sont impliquées dans la mobilité des cellules et des organismes, comme les myosines qui permettent la contraction musculaire, transformant l’énergie chimique en énergie mécanique. Certaines jouent un rôle dans le conditionnement de l’ADN, l’ADN étant enroulé autour de protéines appelées les histones (voir Figure 1.3), d’autres sont impliquées dans la régulation de l’expression génétique, comme les facteurs de transcription accompagnant l’ARN polymérase lors de la transcription. Certaines font office de transporteuses du matériel cellulaire d’un point à un autre, comme la kinésine évoluant sur des structures de microtubules (cf. Figure 1.5).Une protéine est constituée d’une succession d’acides aminés liés entre eux dont il existe 22 sortes différentes. Les acides aminés sont composés d’atomes de carbone, d’hydrogène, d’oxygène et d’azote, certains intégrant aussi un atome de soufre ou de sélénium. Ces acides aminés possèdent une partie commune, le squelette, et une partie spécifique appelée la chaîne latérale, qui caractérise le type d’acide aminé. C’est au niveau de la partie commune que les acides aminés sont liés par une liaison peptidique, la séquence des parties communes constituant la chaîne principale (ou squelette) de la protéine (cf. Figure 1.6a et 1.6b). La chaîne latérale spécifique à chaque type d’acide aminé donne lieu à des propriétés physico- chimiques différentes. Chaque acide aminé peut être représenté par la formule générique H2N-HCR-COOH, dans laquelle R désigne la chaîne latérale (cf. Figure 1.6a).Il est possible de classer les acides aminés selon plusieurs critères, depuis leur taille jusque leur propriété hydrophile (affinité avec l’eau) ou leur polarité. Il existe cependant un classement commun qui les regroupe en six groupes fonctionnels : Les acides aminés aliphatiques (Glycine, Alanine, Valine, Leucine et Isoleucine), les acides aminés avec groupement hydroxyle, sulfurique ou sélénique (Sérine, Thréonine, Méthionine, Cystéine et Sélénocystéine), les acides aminés cycliques (Proline), les acides aminés aromatiques (Phénylalanine, Tyrosine et Tryptophane), les acides aminés basiques (Histidine, Lysine et Arginine) et enfin les acides aminés acides et leurs amides (Aspartate, Glutamate, Asparagine et Glutamine) (voir Figure 1.7). La séquence des acides aminés, pouvant être représentée par une suite de lettres choisies parmi un alphabet de 22 lettres correspondantes chacune à un type d’acide aminé, est appelée la structure primaire d’une protéine. La protéine va adopter, contrainte par les interactions physiques et chimiques entre les différents atomes de la chaîne principale et latérale, des structurations locales particulières. Ces motifs structuraux formés sont au nombre de 3 : hélices, feuillets et coudes et leur enchaînement est appelé la structure secondaire de la protéine (voir Figure 1.8a). Enfin, les protéines possèdent également des motifs plus importants, souvent le résultat de l’agencement dans l’espace des motifs de structures secondaires cités précédemment. C’est la structure tertiaire ou structure tridimensionnelle de la protéine (cf. Figure 1.8b). Cette structuration est due aux interactions proche et longue distance formées par les chaînes latérales des acides aminés. Parmi ces interactions, on retrouve les attractions/répulsions électrostatiques des acides aminés chargés électriquement, l’effet hydrophobe est le phénomène d’enfouissement et de regroupement des régions dont le ratio d’acides aminés hydrophobes est important. Ces régions vont se retrouver à l’intérieur de la protéine alors qu’à l’inverse, les régions dites hydrophiles vont majoritairement se situer en surface de la protéine. Cette structuration, bien que primordiale, n’apparait pas de façon si précise chez toutes les protéines. En effet, certaines d’entre-elles sont désordonnées et ne vont se structurer qu’au contact avec d’autres molécules ou parfois ne jamais adopter de structures précises.
Introduction |