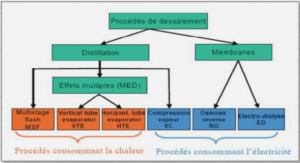
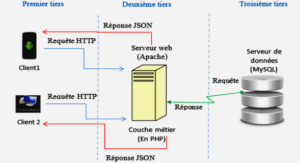
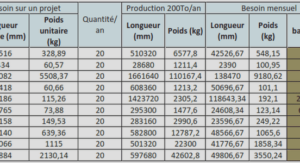
Extraction des protéines du stratum corneum
Pour extraire les protéines, 64mg de poudre acétonique sont réhydratés avec 6,4mL de tampon (PBS, 1M NaCl, 0,1% Triton X100 v/v). Le mélange est homogénéisé dans un potter en verre. Après centrifugation (10min, 10 000g, 4°C), le surnageant est récupéré et passé à travers un filtre 0,45µm puis 0,22µm. La quantité de protéines du surnageant est mesurée par un dosage Bio-Rad. Toutes les étapes de purification ont été réalisées à 4°C sur une station de chromatographie biologique (Bio-Rad). Les extraits de protéines ont été concentrés de 4mL à 1mL par ultrafiltration sur une membrane de seuil 5kDa (Vivaspin 6, Vivascience). L’extrait de protéines a été séparé par chromatographie d’exclusion stérique sur une colonne G75HR10/30, préalablement équilibrée par du tampon de migration (PBS, 1M NaCl, Triton X100 0,1%). La séparation a été réalisée à 400µL/min et le volume des fractions collectées est de 150µL. Les fractions de deux injections successives de 250µL ont été réunies. Un autre extrait de protéines a été séparé par échange de cations sur une colonne Mono S 5/50 GL (Tricorn, GE Healthcare) utilisée en tampon acétate 50mM pH=3,5 (tampon A ; tampon B=tampon A + 1M NaCl). Avant injection, le tampon de l’extrait a été remplacé par du tampon A avec une cartouche EconoPack 10DG (Bio-Rad) : 9mL d’extrait protéique ont été chargés et élués avec 12mL de tampon A. 11mL ont été injectés sur la colonne d’échange de cations. La séparation est réalisée avec un gradient linéaire de 0 à 1M NaCl à 300µL/min en 50min. Le volume des fractions collecté est de 450µL. Le kit d’essai Enzcheck (Molecular Probes) contenant de la caséine BODIPY FL comme substrat générique a été utilisé en suivant la procédure du fabricant, i.e. en maintenant un rapport extrait/substrat dilué de 1:10 (v/v). L’incubation dure une nuit à 37°C. Cette étude a été faite à différents pH : tampon acétate 0,1M pH=5 et tampon Tris 0,1M pH=8. La fluorescence du substrat est mesurée avec une longueur d’onde d’excitation à 485nm et d’émission de 535nm. Les substrats S2288 et S2586 ont été utilisés respectivement pour tester l’activité des sérine protéases (et plus particulièrement de la trypsine) et de la chymotrypsine. S2288 a été employé à une concentration finale de 2,5nmol/µL et avec un rapport extrait/substrat dilué de 5:200 (v/v). S2586 a été utilisé à une concentration finale de 250pmol/µL et avec un rapport extrait/substrat dilué de 5:200 (v/v). L’incubation dure une nuit à 37°C à pH=8. L’absorbance est mesurée à la longueur d’onde de 405nm. Pour les tests enzymatiques spécifiques de la cornéodesmosine, 13 peptides synthétiques (JPT Peptides Technologies, Allemagne) ont été utilisés à la concentration finale de 10pmol/µL et avec un rapport extrait/substrat dilué de 1:20 (v/v). L’incubation à 37°C a duré au moins une nuit et l’étude a été faite à la fois à pH=5 et pH=8 (cf 0,3µL d’échantillon (mélange dessalé d’un peptide CDSN avec ou sans extrait de protéases) ont été déposés avec 0,6µL de matrice CHCA (4mg/mL, 6:4:0,01 ACN/H2O/TFA v/v/v, 10mM citrate d’ammonium). Les analyses MS ont été réalisées sur un spectromètre de masse MALDI-TOF/TOF 4800 (Applera Applied Biosystems, Framingham, MA) en mode réflectron positif (gamme de masse 500-2 000), intensité du laser 2 100, 1 000 tirs cumulés). Les données MS/MS ont été acquises en utilisant de l’air comme gaz de collision et avec une énergie de 2keV (fenêtre de sélection de 5Da, intensité du laser 4 500, 2 400 tirs cumulés). L’étalonnage est obtenu de façon externe en réalisant un modèle de plaque à partir de 13 spots sur lesquels est déposé un mélange de peptides standards (Proteomix Peptide calibration Mix 4, LaserBio Labs, Sophia Antipolis, France) contenant le fragment 1- 5 de la bradykinine (573,315Da), l’angiotensine humaine II (1 046,542Da), la neurotensine (1 672,918Da), le fragment 18-39 de l’ACTH (2 464,199Da) et la chaîne B de l’insuline (3 494,651Da).