Analyse et modélisation du repliement spatial de
l’épigénome
Résultats : Applications du modèle
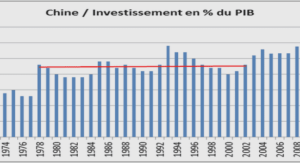
Dans cette sous section nous allons illustrer à partir de notre modèle le comportement générique à l’équilibre d’un homopolymère, d’un hétéropolymère simple composé de deux types de blocs et d’un hétéropolymère dont l’état des blocs est défini selon l’épigénome de la drosophile. Les polymères seront constitués de 120 monomères de 10kb, soit 1.2Mb. Le fait d’étudier des bouts de 1.2Mb n’est pas si déraisonnable qu’il peut y paraître puisque l’on a introduit dans la partie 3.1.3 que plus de 50% des contacts intra chromosomiques sont détectés pour des distances génomiques inférieures à 1Mb et puisque l’on introduit dans notre modèle un champ générique rendant compte de l’environnement de manière effective. Pour les différents hamiltoniens définis en section 4.2.3, on résoudra le système d’équations différentielles défini par l’équation 4.18 dans le cas stationnaire, donc avec ζ dDij dt = 0. Cette résolution est réalisée avec l’algorithme itératif ou dans le cas où celui-ci ne converge pas avec l’algorithme de Runge-Kutta.Comme expliqué dans la section précédente, la matrice des distances carrés moyennes D à l’état stationnaire obtenue après intégration de l’équation 4.18, nous donne la possibilité de générer des configurations typiques (cf. 4.2.1.5) et nous permet de calculer la matrice des
Approche gaussienne auto-cohérente
Figure 4.2.14 – Exemple de résolution du système 4.18 pour le copolymère (A10B10) avec deux algorithmes différents. En haut à gauche : matrice des potentiels d’interaction U caractérisant le polymère étudié ici. En bas à gauche : matrice des distances initiales choisie pour débuter la résolution de l’équation 4.18. À droite : évolution de l’écart entre la matrice Dk calculée lors de l’itération k et la matrice Dk−1 calculée lors de l’itération précédente (Éq. 4.41) en fonction de l’itération k et dans le cas de la résolution avec la méthode de Runge-Kutta et avec l’algorithme itératif (λ = 0.025) (resp. en bleu et en rouge). On peut voir en insert des exemples de matrices Dk correspondant aux itérations k = 10, k = 100 et k correspondant à la dernière itération. L’algorithme itératif et celui de
Runge-Kutta ont respectivement convergé en 3000s et 3s, donc une différence d’un facteur 1000 pour ce cas précis. contacts, C. Cette conversion de C à D se fait avec la relation 4.13. Dans tout cette section « Résultats », la constante A impliquée dans cette équation 4.13 est calculée en imposant arbitrairement que la distance moyenne entre deux monomères plus proches voisins soit de 1. Cette condition implique qu’en moyenne pour tout monomère i, Di i+1 ≈ 1/3 puisque pour rappel, Dij =D(Xi − Xj)2E/3). La constante est donc A =1NPi Ci i+1(1/3)−3/2. Comme C et D sont
donc liés par une relation mathématique claire, on ne présentera dans cette partie que les matrices C, et pas D afin d’éviter une redondance.
Conditions initiales et multistabilité mathématique
Nous avons vu dans la partie 4.2.4 que la résolution du système 4.18 nécessite au départ une matrice des distances initiales, Dinitiale. Afin de sonder l’éventuelle existence de plusieurs points fixes pour le système dynamique 4.18, on débutera alors systématiquement la
4. Modélisation de la chromatine par un copolymère par bloc
De gauche à droite, carte des contacts, C, (avec même échelle de couleur pour toutes), probabilité de contact Pc en fonction de la distance génomique s et exemple d’une structure obtenue avec l’approximation gaussienne. De haut en bas, les quatre configurations choisies comme état initial : polymère gaussien sous forme de pelote, polymère dans sa phase générique (construit à partir d’une probabilité de contact évoluant en s−1), polymère dans un état microphasé et polymère issu d’un cas expérimental. résolution avec quatre conditions initiales très différentes : polymère dans sa phase gaussienne, générique, microphasée et un cas expérimental (Fig. 4.2.15). Ces matrices Dinitiale sont construites avec le formalisme auto-cohérent. Concrètement, il s’agit de résoudre l’équation 4.18 pour obtenir les matrices Dinitiale recherchées en commençant, pour ces cas là, la résolution avec comme matrice des distances initiales, une matrice gaussienne calculée avec l’équation 4.7 qui relie D et K
−11 avec la matrice K1 donnée par la formule 4.6. Pour obtenir la matrice Dinitiale correspondant au polymère sous forme de pelote, on résout le système avec
l’hamiltonien H2A et une matrice d’interaction entre monomères totale U = Uns + Us = 0. Pour obtenir la forme générique, on résout 4.18 avec H2I et U = 0. Pour la microphase, on utilise H2A et U = Uns avec Unsij = −6δeiej avec δeiej = 1 si i et j sont dans le même état épigénomique. Enfin, la dernière matrice initiale a été obtenue à partir de la carte de contact
expérimentale de la drosophile entre 12.5 et 13.7Mb [Sexton et al., 2012]. La transformation de la carte de contact en carte de distance s’est faite avec la relation 4.13.Pour un copolymère donné, on comparera les quatre solutions obtenues que l’on note D1,D2, D3, D4 en calculant l’écart ǫDiDj entre chaque couple de solution (Éq. 4.45).Après observation de plusieurs exemples, et de manière cohérente avec la précision donnée par les algorithmes de résolution (cf. section 4.2.4 et en particulier Fig. 4.2.12), on choisit de dire que deux matrices solutions Di et Dj sont identiques si l’écart entre elles est inférieur à 10−3 Dans le cas où, pour le même système 4.18, des solutions différentes sont trouvées selon la condition initiale utilisée, on dira que le système présente une multistabilité mathématique (c’est-à-dire l’existence de plusieurs points fixes pour le système dynamique 4.18). On considère que si, ǫ4, le maximum des écarts calculés, (ǫ4 = maxi,j∈J1,4.
1 Introduction |