Sommaire: Développement d’une PCR en temps réel pour la détection des brucella et relations avec le genre ochrobactrum
RESUME
SUMMARY
LISTE DES ABBREVIATIONS
I. INTRODUCTION BIBLIOGRAPHIQUE
A. Brucella et Brucellose
1. Introduction
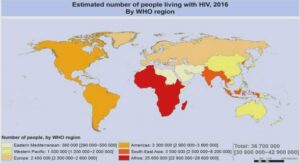
2. Données épidémiologiques

a) Situation mondiale
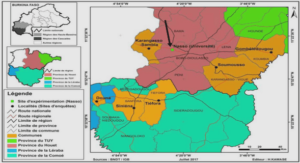
b) Situation en méditerranée
c) Situation en France
3. Pathogénie et aspects cliniques
4. Le genre Brucella
a) Les alpha-proteobacteries
b) Description du genre Brucella
c) Taxonomie et hôte
d) Le génome
e) Antigènes
B. Relation entre Brucella et Ochrobactrum
1. Présentation du genre Ochrobactrum.
a) Description
b) Taxonomie
c) Manifestations cliniques
2. Relation entre Brucella et Ochrobactrum
C. Diagnostic des Brucelloses animales
1. Généralités
2. Diagnostic bactériologique
3. Diagnostic sérologique de Brucella
4. Diagnostic moléculaire
D. Gènes et protéines d’intérêt de l’étude
1. Gène bcsp31
2. Séquence d’insertion IS711 et séquence répétées BRU-RS1, BRU-RS2
3. Protéines membranaires : Omp10, Omp16 et Omp19
4. Protéine périplasmique : BP26
5. Gène pérosamine synthétase (per)
E. But de l’étude
II. RESULTATS
A. Résultats de la 1ère partie : Développement d’un outil de diagnostic du genre Brucella basé sur la PCR temps réel
1. Détection des Brucella par la technique de PCR conventionnelle et en temps réel
a) Choix des cibles
b) Etude d’exclusivité
c) Etude d’inclusivité
d) Limite de détectabilité
e) Répétabilité et reproductibilité
2. Choix et validation de la PCR en temps réel sur des échantillons biologiques
a) Etude préliminaire : échantillons de rates de sangliers et d’animaux expérimentalement infectés
b) Echantillons biologiques d’animaux négatifs
c) Echantillons biologiques d’animaux positifs
d) Analyse de la sensibilité et de la spécificité diagnostique
e) Détermination du seuil de positivité en PCR temps réel
B. Résultats de la 2ème partie : Etude phylogénétique entre le genre Brucella et le genre Ochrobactrum
1. Recherche de la séquence répétée BRU-RS et de la séquence d’insertion IS711 chez Ochrobactrum
2. Détection de séquences homologues à celles des gènes omp10, omp16, omp19 et bp26 de Brucella spp. dans un panel d’espèces d’Ochrobactrum
3. Recherche d’épitopes communs sur un panel d’espèces Ochrobactrum par utilisa-tion d’anticorps spécifiques dirigés contre Omp10, Omp16, Omp19 et BP26 de Brucella spp
III. DISCUSSION
IV. CONCLUSION GENERALE ET PERSPECTIVES
V. MATERIELS ET METHODES
A. Matériel biologique utilisé
1. Souches Brucella
2. Souches Ochrobactrum
3. Bactéries ayant un lien avec Brucella
4. Autres bactéries
5. Echantillons biologiques
B. Techniques bactériologiques
1. Souches Brucella
2. Souches Ochrobactrum
3. Autres bactéries
4. Echantillons biologiques
C. Préparation de l’ADN
D. Amorces et sondes
E. Technique de PCR conventionnelle simple et double amplification
F. Technique PCR en temps réel
1. Technique PCR en temps réel
2. Répétabilité et reproductibilité
G. Sensibilité et spécificité analytique
H. Choix du seuil de détection en PCR temps réel
1. Estimation de la sensibilité et de la spécificité diagnostique
2. Courbe ROC
3. Indice de Youden
4. Comparaison de la moyenne des différence : « le test de t apparié »
5. Comparaison de 2 moyennes observées
6. Comparaison de pourcentages de réponses positives de 2 séries appariées
I. Technique du Southern Blot
J. ELISA
1. Inactivation des bactéries
2. Choix des anticorps
3. Technique ELISA
VI. REFERENCES BIBLIOGRAPHIQUES
VII. COMMUNICATION AFFICHEE ET PUBLICATION
VIII. ARTICLE
IV. ANNEXE
Annexe 1 : Espèces et biovars de Brucella utilisés dans l’étude
Annexe 2 : Souches Ochrobactrum utilisées dans l’étude
Annexe 3 : Micro-organismes non-Brucella utilisés pour l’étude de spécificité
Annexe 4 : Préparation de milieux de culture
Annexe 5 : Préparation d’ADN génomique par la méthode phénol-chloroforme
Annexe 6 : Protocole d’extraction : kit Macherey Nagel (Genomic DNA from tissue)
Annexe 7 : Protocole d’extraction : kit Roche (High Pure PCR Template Preparation)
Annexe 8 : Protocole d’extraction : kit BioRad (InstaGene Matrix)
Annexe 9 : Protocole d’extraction : kit manuel Qiagen (DNeasy Blood & Tissue)
Annexe 10 : Protocole d’extraction : kit robot Qiagen (MagAttract® DNA Mini M48, BioRobot® M48 workstation)
Extrait du mémoire développement d’une PCR en temps réel pour la détection des brucella et relations avec le genre ochrobactrum
I. INTRODUCTION BIBLIOGRAPHIQUE
A. Brucella et Brucellose
1. Introduction
Au cours de son histoire, la brucellose s’est vu donnée plusieurs noms comme fièvre de malte, de Chypre, de Gibraltar (pour les hommes), fièvre méditerranéenne, abortive, ondulante, sudoro-algique, ou encore Mélitococcie, maladie de Bang, septicémie de Bruce, avortement épizootique ou contagieux (pour les animaux) ou encore épididymite contagieuse du bélier (chez les ovins) (Souk-Aloun, 1989).
La première description clinique de la maladie a été publiée par Martson en 1859 sous le nom de fièvre méditerranéenne (Roux, 1989) et présentait la maladie comme fébrile et ondulante.
Mais c’est en 1887 que l’agent responsable de la maladie est isolé par David Bruce, médecin militaire anglais, à partir de la rate d’un soldat décédé de la maladie. Il observe avec l’aide d’un microscope un grand nombre de bactérie. Ce germe reçoit alors l’appellation de « Micrococcus melitensis » (Roux, 1989).
En 1897, Bang, un vétérinaire danois, isole un bacille de produits d’avortements bovins qu’il appelle Bacillus abortus bovis ; et Wright met au point le premier test diagnostique sérologique qui porte son nom : réaction d’agglutination de Wright.
En 1905, le bactériologiste maltais Zamitt découvre par la réaction d’agglutination de Wright que les chèvres de l’ile de Malte infectent les hommes par le lait et constituent ainsi un réservoir de la maladie.
En 1914, Traum isole une bactérie semblable à celle isolée par Bang, à partir de fœtus porcin et la nomme Bacillus abortus suis. Quatre ans plus tard, une bactériologiste américaine, Alice Evans, propose une parenté entre ces deux micro-organismes. C’est ainsi que le genre Brucella est établi par Meyer et Shaw en 1920 (Roux, 1989) en l’honneur de David Bruce.
En 1929, Huddleson développe des méthodes bactériologiques permettant de distinguer les espèces B. melitensis, B. abortus et B. suis.
En 1930, aux Etats Unis, Buck démontre le pouvoir protecteur de la souche non virulente Brucella abortus S19 sur des bovins qui sera plus tard utilisée comme vaccin pour prévenir la brucellose bovine (Schurig et al., 2002). En 1932, Wilson et Miles affirment la présence d’antigènes de structure lipopolycaccharidique chez Brucella, appelés A et M et inégalement répartis selon les espèces.
La souche B. abortus 45/20 est testée pour la première fois comme vaccin en 1938 par Mc Ewen et Priestley ; les travaux cumulées de Edwards et collaborateurs et de Taylor et collaborateurs, en 1945 et 1949 en font la première souche vaccinale inactivée mise au point (Schurig et al., 2002). La première souche vaccinale vivante atténuée, B.melitensis Rev1, est développée par Elberg et Faunce en 1957 (Schurig et al., 2002).
…………
Si le lien ne fonctionne pas correctement, veuillez nous contacter (mentionner le lien dans votre message)
Développement d’une PCR en temps réel pour la détection des brucella et relations avec le genre ochrobactrum (2.46 MB) (Rapport PDF)