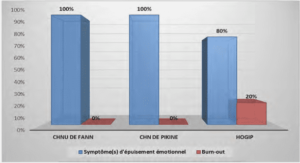
Télécharger le fichier original (Mémoire de fin d’études)
Mécanismes de résistance
Les bactéries ont développé différents mécanismes afin de neutraliser l’action des agents antibactériens, les plus répandus étant :
l’inactivation enzymatique de l’antibiotique la modification ou le remplacement de la cible de l’antimicrobien l’efflux actif la diminution de la perméabilité de la membrane D’autres mécanismes tels que la protection ou la surproduction de la cible de l’antibiotique sont également décrits. Ils sont cependant plus rares.
Inactivation enzymatique de l’antibiotique
L’inactivation enzymatique de l’antibiotique représente le principal mécanisme de résistance des bêta-lactamines, des aminoglycosides et des phénicolés. On décrit également ce type de résistance pour le groupe MLS (macrolides, lincosamides, streptogramines), pour les tétracyclines, pour la fosfomycine et plus récemment pour les fluoroquinolones, bien que cette inactivation ne représente pas le mécanisme de résistance qui prévaut pour ces molécules. L’enzyme en modifiant le noyau actif de l’antibiotique par clivage ou par addition d’un groupement chimique, empêche la fixation de l’antimicrobien sur sa cible et provoque une perte d’activité.
Inactivation enzymatique des bêta-lactames
La production de bêta-lactamase est un mécanisme que l’on retrouve aussi bien chez les bactéries à Gram positif que à Gram négatif, il s’agit du mode de résistance le plus courant. Le support génétique qui code pour ces enzymes est soit d’origine plasmidique soit chromosomique. Les bêta-lactamines sont des enzymes d’inactivation de type sérine (classes A, C et D) ou métallo enzymes (classe B) dont les substrats sont des bêta-lactamines et qui peuvent être classées en sous-groupes selon la structure du noyau de base (péname, oxapéname, pénème, céphéme, oxacéphème, azétididone). Compte tenu de l’extrême diversité de ce groupe enzymatique, les besoins d’une classification sont anciens. La première classification basée sur des critères scientifiques a été proposée dans les années 75 par Ambler [9], elle prend en compte les analogies de séquence peptidique, en particulier celles du site enzymatique. Ainsi, quatre classes (A, B, C et D) ont été identifiées. La classification fonctionnelle de Bush, Jacoby, Medeiros [10, 11] reflète mieux le spectre exact des enzymes, prenant en compte le profil du substrat (pénicilline, oxacilline, carbénicilline, céphaloridine, C3G, imipénème), ainsi que le profil d’inhibition. Ainsi apparait la notion de groupe fonctionnel tel le groupe 2b qui se subdivise en sous-groupes 2ba, 2bc… Mais ce type d’enzyme a un potentiel évolutif et une seule mutation (ponctuelle) peut changer le profil d’inactivation et celui d’inhibition : groupe 2b se subdivise alors en 2be. Néanmoins, elle est peu utilisée en pratique médicale [10, 11, 12].
Inactivation enzymatique des aminosides
L’inactivation enzymatique des aminosides est le mécanisme de résistance le plus souvent observé. Il permet d’expliquer la résistance de plus de 95% des souches d’entérobactéries résistantes aux aminosides, de 95% des souches d’Acinetobacter spp, de 50% des souches de Pseudomonas aeruginosa et de 95% des souches de bactéries à Gram positif [13, 14]. Tous les aminosides possèdent des groupements aminés et des groupements hydroxyles nécessaires à leur activité et ces groupements peuvent être la cible de trois classes d’enzymes. Les phosphotransférases ou APH transfèrent, un groupement phosphate sur les groupements hydroxyles, les nucléotidyltransférases ou O-adénylyl (ANT ou AAD) agissent par adénylations des groupements hydroxyles, lesn acétyltransférases ou AAC catalysent l’acétylation des groupements aminés.
Il faut noter les points suivants :
Un seul aminoside peut être inactivé par plusieurs enzymes Une seule enzyme peut inactiver plusieurs antibiotiques Une seule souche peut produire plusieurs enzymes.
Inactivation enzymatique des phénicolés
Pour le chloramphénicol et le thiamphénicol, l’inactivation enzymatique est le mécanisme de résistance le plus fréquent. Elle agit par acétylation par une 10 chloramphénicol acétyltransférase du groupement hydroxyle de la molécule. On a identifié 3 enzymes chez les bactéries à Gram négatif et cinq chez les bactéries à Gram positif [18, 19]. À l’exception de Streptococcus pneumoniae, ces enzymes sont codées par des plasmides. Les chloramphénicols acétyltransférases sont cependant inactifs sur le florfénicol.
L’efflux actif
Ce sont des mécanismes de transport membranaire universellement répandus chez des organismes vivants. Ils ont un rôle clé dans la physiologie bactérienne : (Préserver l’équilibre physico-chimique du milieu intracellulaire en s’opposant à l’accumulation de substances naturelles ou synthétiques toxiques, transport de substances nutritives et export de substances toxiques.) Le mécanisme de résistance par le système des efflux réside dans l’excrétion active de l’antibiotique par les pompes à protons, il s’agit là d’un mode de résistance intrinsèque des bactéries, toutefois l’exposition aux antibiotiques entraîne la surexpression par mutation de transporteurs, ce qui entraîne une hausse des résistances bactériennes qui peut être simultanée à des antibiotiques non reliés structuralement [20, 21]. On différencie les pompes à efflux par :
spécificité ou non des molécules exportées structure : une à trois protéines type d’énergie nécessaire : ATP ou force proton-motrice mode expression : inductible ou constitutif. Il existe cinq grandes familles des systèmes d’efflux actif
ATP binding cassette transporter
12 domaines transmembranaires et un domaine de fixation d’ATP
RND : resistance nodulation cell division avec trois composants : Protéine de transport dans la membrane cytoplasmique Protéine dans le périplasme formant un canal reliant les deux membranes Protéine dans la membrane externe type porine expulsant le substrat MFS ou MF: major facilitator superfamily : 12 ou 14 domaines transmembranaires
SMR : Small multidrug resistances : 4 domaines transmembranaires MATE : multidrug and toxic exclusive
Diminution de la perméabilité de la membrane
Pour agir, les antibiotiques doivent pénétrer dans la cellule bactérienne. Beaucoup d’antibiotiques utilisent les systèmes de transport propres à la bactérie pour ses échanges avec l’extérieur pour entrer [22]. Pour résister, la bactérie contrecarre cette entrée de toxiques en diminuant la perméabilité de sa membrane par :
Une altération des porines : ce mode de résistance n’affecte que les bactéries Gram négatif. Chez ces bactéries, la membrane externe constitue une barrière de diffusion très efficace. L’antibiotique ne peut traverser cette barrière qu’en empruntant des structures particulières : les porines (protéines formant les pores de la membrane). Le passage des antibiotiques à travers les porines est d’autant plus facile que les molécules sont de petite taille, neutres et très hydrophiles. Toute modification des porines rend le passage des molécules hydrophobes (comme la famille des béta-lactamines) encore plus difficile. L’absence de passage ou l’augmentation du temps de passage protège les bactéries et les rend résistantes. Une inhibition du transport actif Une inhibition de la pénétration à travers les peptidoglycanes recouvrant la membrane plasmique chez les bactéries Gram positif. La modification de la composition du lipopolysaccharide (LPS), soit dans le polysaccharide, soit dans le core, peut aussi être à l’origine d’une diminution de la perméabilité.
Modification ou remplacement de la cible de l’antibiotique
Les glycopeptides (vancomycine, et teicoplanine) ont une affinité pour les précurseurs du peptidoglycane comportant le dipeptide D-alanyl-D-alanine. Les cibles potentielles sont donc soit intra cytoplasmiques soit situées au niveau de la paroi en formation. Ces cibles ne sont pas toutes atteintes, car elles ne sont pas toutes accessibles aux glycopeptides. Aucune cible n’est atteinte chez les bactéries à Gram négatif, car ces antibiotiques ne peuvent pas traverser la membrane externe. Ceci explique que les glycopeptides ont un spectre étroit limité aux bactéries à Gram positif (principalement streptocoques, entérocoques et staphylocoques). Chez les bactéries à Gram positif, ces antibiotiques diffusent librement à travers les mailles du peptidoglycane. En revanche, ils ne peuvent traverser la membrane cytoplasmique et leur action s’exerce sur la paroi en formation.
Table des matières
INTRODUCTION
PREMIERE PARTIE : RAPPEL
CHAPITRE I : RESISTANCE AUX ANTIBIOTIQUES
I. Définition
II. Mécanismes de résistance
1. Inactivation enzymatique de l’antibiotique
1.1. Inactivation enzymatique des bêta-lactames
1.2. Inactivation enzymatique des aminosides
1.3. Inactivation enzymatique des phénicolés
2. L’efflux actif
3. Diminution de la perméabilité de la membrane
4. Modification ou remplacement de la cible de l’antibiotique
4.1. Altération des précurseurs de la paroi bactérienne
4.2. Modification d’affinité de la cible
4.3. Substitution de cible
III. Antibiogramme
1. Méthodes
1.1. Méthode de dilution : détermination de la CMI
1.2. Méthode de diffusion
2. Principe et procédé de l’antibiogramme
3. Interprétation de l’antibiogramme
CHAPITRE II : LES BACTERIES MULTI RESISTANTES
I. Définition
II. Les principales bactéries multi résistantes
1. Staphylococcus aureus résistant à la méticilline
2. Entérobactéries productrices de béta lactamases à spectre élargi
2.1. Définition
2.2. Historique des BLSE
2.3. Méthodes de détection phénotypique de la production de BLSE
3. Entérobactéries productrices de carbapénémases
3.1. Classification des carbapénémases
3.2. Méthodes de détection des entérobactéries productrices de carbapénémases
4. Acinetobacter baumannii résistant à l’Imipénème
4.1. Mécanismes enzymatiques
4.2. Mécanismes non-enzymatiques
5. Pseudomonas aeruginosa résistant à la Céftazidime
6. Entérocoque résistant à la vancomycine
CHAPITRE III : EPIDEMIOLOGIE DES BACTERIES MULTI RESISTANTES
Facteurs de risques de la multi résistance
II. Evolution en Afrique de l’ouest
1. Entérobactéries productrices de Bêta-lactamases à Spectre Elargi (BLSE)
2. Entérobactéries productrices de carbapénémases
3. Staphylococcus aureus résistant à la méticilline
III. Contexte épidémiologique de la multi résistance au Sénégal
CHAPITRE IV : STRATEGIES ET MESURES DE LUTTE CONTRE LES BACTERIES MULTI RESISTANTES
Stratégies et mesures de lutte
1. Identification des patients porteurs de BMR
2. Méthodes d’isolement
2.1. Isolement technique
2.2. Isolement géographique
II. Mesures complémentaires
DEUXIEME PARTIE : TRAVAIL PERSONNEL
CHAPITRE I : CADRE DE L’ETUDE
CHAPITRE II : MATERIEL ET METHODES
I. Type et période d’étude
II. Méthodes
1. Recueil de données
2. Exploitation des données
3. Méthode de l’antibiogramme
CHAPITRE III : RESULTATS
I. Caractéristiques épidémiologiques
II. Aspects bactériologiques des infections à BMR
1. Sensibilité des entérobactéries sécrétrices de bétalactamases à spectre élargi aux autres familles d’antibiotiques
2. Sensibilité des staphylocoques résistants à la méticilline aux antibiotiques
3. Sensibilité des bacilles non fermentaires aux antibiotiques
4. Sensibilité de Streptococcus spp vis-à-vis des antibiotiques testés
CHAPITRE 4: DISCUSSION
I. Aspects épidémiologiques
II. Aspects bactériologiques
CONCLUSION ET RECOMMANDATIONS
REFERENCES BIBLIOGRAPHIQUES