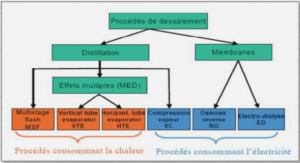
Synthèse d’observateurs pour une culture bactérienne en batch avec recyclage de substrat
Dans ce chapitre, nous rappelons le modèle mathématique qui décrit la dynamique de la croissance d’une biomasse de concentration X sur un substrat de concentration S en culture batch tout en tenant compte de l’accumulation des cellules non viables (X ). Le recyclage d’une partie des cellules mortes en substrat, en fonction du temps t est également considéré.Nous faisons face à un modèle pour lequel nos paramètres ne sont pas identifiables à l’équilibre. Ainsi, on ne peut pas appliquer simplement les techniques classiques d’identification, qui exigent habituelle- ment l’observabilité globale du système. En outre, nous comptons reconstruire les variables d’état non mesurées en ligne (concentrations des cellules viables et non viables), aussi bien que les paramètres. Pour cela, nous proposons un couplage de deux observateurs non linéaires en cascade avec différentes échelles de temps, fournissant une convergence pratique de l’erreur d’estimation.Il s’agit d’une extension du modèle de Monod. Le paramètre m > 0 est le taux de mortalité que l’on suppose significatif lorsque µ(S) prend de petites valeurs, δ ∈ (0, 1) décrit la part des cellules mortes non éclatées, λ > 0 est le facteur de recyclage, Y est le coefficient de rendement de la conversion de substrat en biomasse. D’un point de vue biologique, on suppose que λ est plus petit que le rendement de croissance :H1. Monod sont connus et que le but est d’identifier les paramètres m, δ et λ , et de reconstruire en ligne les variables d’état X et X . Sans perte de généralité, on suppose que le taux de croissance µ(·) est une fonction vérifiant les hypothèses suivantes : , en se basant sur les mesures disponibles. On peut immédiatement voir que les paramètres (m, a, k) ne peuvent pas être reconstruits si le système est à l’équilibre. Néanmoins, en dérivant les sorties (y) : jusqu’au le temps τ, il doit, donc, être intégré sur [0, min(τ(t),t)] lorsque le temps actuel est t.
Ces estimations sont utilisées, en ligne, par le second observateur, avec un gain θ = 2. Sur les Figures 3.5, on voit bien que l’erreur d’estimation est petite si l’erreur d’estimation donnée par le premier observateur est petite elle aussi. En présence de bruit à basse fréquence (ce qui peut être le cas des applications biologiques), on obtient une bonne robustesse pour les estimations des paramètres α et β et des variables d’état x et x . L’estimation du paramètre m est plus affectée par le bruit. Ceci peut être expliqué par la structure de l’équation (3.5) : l’estimation de m est reliée au dérivées secondes des deux observations y L’extension du modèle de Monod avec un compartiment additionnel pour les cellules mortes et un terme de recyclage de substrat n’est pas identifiable en considérant les observations de la concentration en substrat et de la biomasse totale. Néanmoins, nous avons prouvé que le modèle peut être écrit sous une forme particulière cascade, en considérant deux échelles de temps.Cette décomposition permettre la conception, séparément, de deux observateurs, puis les coupler en- semble en cascade. Le premier est à échelle de temps bornée, expliquant pourquoi le système n’est pas identifiable à l’équilibre, alors que le second est à échelle de temps non bornée. Cette construction four- nit une convergence pratique des observateurs couplés.Les deux observateurs sont à grand-gain inspirés de [37] avec une construction explicite de l’exten- sion Lipschitzienne de la dynamique (cf. [78]). D’autres choix d’observateurs pourraient avoir été faits et appliqués à cette structure particulière. Une telle décomposition pourrait être appliquée à d’autres systèmes qui sont pas identifiables ou pas observable à l’équilibre.