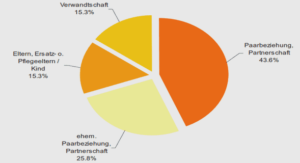
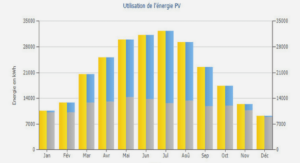
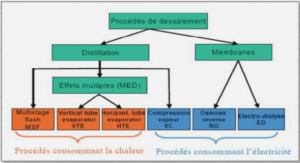
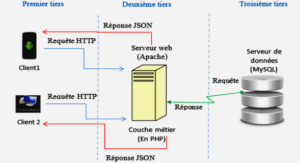
une méthode de distances rapide et précise pour estimer le taux de substitution à partir d’un ensemble de séquences hétérochrones
Nous présentons ici une méthode de distances, Ultrametric Least Squares (ULS), qui es- time le taux de substitution d’un ensemble de séquences hétérochrones, en faisant l’hy- pothèse d’une horloge moléculaire stricte. Cette méthode corrige la distance évolutive entre séquences, par l’adjonction d’un facteur correctif aux séquences non contempo- raines. Ce facteur est proportionnel au taux de substitution à estimer, ainsi qu’à l’ancien- neté de la séquence en question. Le taux de substitution est alors estimé par la minimisa- tion d’un critère quadratique, qui mesure l’ultramétricité de la distance corrigée. Nous montrons que ce critère est parabolique par morceaux, et proposons un algorithme effi- où est le nombre de séquences, pour minimiser ce critère. Nous montrons aussi qu’il est possible de borner cette complexité et sans perte de précision par un tirage aléatoire de triplets. Notre méthode peut être étendue à l’estimation de plu- sieurs taux de substitution variant au cours du temps, par exemple pour prendre en compte la prise d’un traitement et sa date de début, ou par lignage (horloges molécu- laires locales). ULS est confrontée sur données simulées à d’autres méthodes de dis- tances, comme sUPGMA ou TREBLE, aux régressions linéaires Root-to-Tip et Pairwise- Distance, ainsi qu’à l’approche probabiliste développée dans le logiciel BEAST, qui est à l’heure actuelle considérée comme l’une des plus précises mais est handicapée par un temps de calcul très important. Les expériences montrent qu’ULS est plus précise ou aussi précise que les autres méthodes de distances et que BEAST, tout en étant extrê- mement rapide. Nous présentons ensuite une application d’ULS sur deux jeux de don- nées du VIH.
Les méthodes de distances définissent avec les méthodes probabilistes et les méthodes de parci- monie les trois approches principales permettant l’inférence de phylogénies moléculaires (cf. Cha- pitre 1). Un des principes souvent utilisé avec les méthodes de distances est celui des moindres car- rés (en anglais Least Squares) qui compare les distances évolutives estimées entre paires de sé- quences, contenant des erreurs dues à l’échantillonnage et inhérentes au modèle d’évolution, aux distances patristiques (ou distances de chemin) calculées dans l’arbre estimé (Felsenstein, 1997; Bulmer, 1991; Fitch & Margoliash, 1967). Ce principe est non seulement rapide en temps de calcul, mais augmente en précision au fur et à mesure que les erreurs d’estimation dans les distances ten- dent à disparaître. En pratique, il est impossible d’estimer les vraies distances évolutives puisque les modèles d’évolution font des hypothèses simplificatrices, comme, par exemple, l’indépendance des sites. Pour contrer cet effet, plus marqué sur les grandes distances que sur les petites, les méthodes de moindres carrés utilisent généralement une valeur de pondération devant chaque terme de la somme, qui est inverse à la variance de l’estimateur et donc plus faible pour les grandes distances. Ainsi, les méthodes de moindres carrés pondérées Weighted Least Squares, WLS, généralisent la Cette approche de pondération est peu exploitée (hormis TREBLE) par les méthodes de distances qui permettent d’estimer le taux de substitution, alors qu’elle est presque universelle pour les mé- thodes d’inférence phylogénétique. À notre connaissance, la méthode sUPGMA est la seule qui uti- lise le principe des moindres carrés, mais sans valeurs de pondération (OLS) (Rodrigo et al, 2007; Drummond & Rodrigo, 2000).
Nous présentons dans ce chapitre une méthode de distances, Ultrametric Least Squares (ULS), qui estime le taux de substitution d’un ensemble de séquences hétérochrones sous l’hypothèse d’une horloge moléculaire stricte, c’est-à-dire sous les hypothèses du modèle SRDT. Cette méthode utilise des triplets de séquences et des pondérations, comme TREBLE, mais propose un algorithme radica- lement différent où on optimise un critère global dont nous montrons qu’il est parabolique par mor- ceaux. Cette méthode est ensuite étendue aux modèles MRDT et DR (mais avec des horloges molécu- laires locales). Les performances de cette méthode sont simultanément comparées avec celles des autres méthodes de distances et celles de la méthode probabiliste de référence, BEAST.