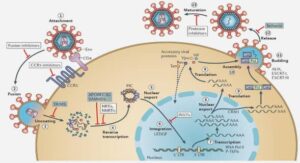
Segmentation d’IRM Cérébrales Multi-Séquences
Segmentation en tissus
Suite aux différents prétrairements présentés dans le chapitre précédent, l’ensemble des images a donc été placé dans un repère spatial unique, à savoir le repère de l’atlas statistique pour permettre l’utilisation d’un a priori sur les labélisation. Le problème majeur est donc maintenant de segmenter les images, et ce afin d’obtenir une cartographie des lésions éventuelles de matière blanche. Il ne faut pas perdre cet objectif de vue : même si la segmentation en tissus que ce chapitre va développer est une étape cruciale dans le processus de détection des lésions, elle n’est pas un but en soi. Dans le choix de la technique, il faudra donc un modèle particulièrement robuste à la qualité des images et à la présence de points aberrants – les lésions de SEP – mais aussi un modèle qui fournit des informations directement exploitables dans la détection et le contourage des lésions.
Choix de la méthode
Le but principal du processus de segmentation est le partitionnement d’une image en régions – également appelées classes – homogènes du point de vue d’un certain nombre de caractéristiques ou critères. En imagerie médicale, la segmentation est très importante, que ce soit pour l’extraction de paramètres ou de mesures sur les images, ainsi que pour la représentation et la visualisation. Beaucoup d’applications nécessitent une classification des images en différentes régions anatomiques : graisse, os, muscles, vaisseaux sanguins, alors que d’autres recherchent une détection des zones pathologiques, comme les tumeurs, ou des lésions cérébrales de matière blanche. De nombreux articles et livres [155, 10, 120] font le résumé des différentes techniques de segmentation disponibles pour l’imagerie médicale ; ce paragraphe ne se veut pas la synthèse de toute cette littérature. Les techniques de segmentation peuvent être divisées en classes de multiples manières : la plus commune est de séparer les méthodes de segmentation de régions qui recherchent un critère d’homogénéité au sein d’une zone, et les méthodes de détection de contours, qui se concentrent sur l’interface entre ces régions. L’arrivée d’outils mathématiques et la diversité des problèmes posés a cependant souvent brisé cette dichotomie : nous parlerons d’abord des méthodes classiques qui ont initié la segmentation, et poursuivrons par les différents développements plus récents : les approches statistiques, la logique floue, les modèles déformables.
Méthodes classiques
Parmi les méthodes classiques, le seuillage est souvent la plus simple, et rarement la plus efficace. De nombreuses techniques de seuillage ont été développées [72, 153] : certaines sont basées sur une analyse d’histogramme, d’autres sur des propriétés locales, comme la moyenne ou l’écart type. Si le seuillage permet de binariser un résultat final, ce qui est toujours nécessaire, il n’est bien souvent pas suffisant pour obtenir une segmentation acceptable, bien que de récentes applications utilisent un seuillage adaptatif local, notamment sur des images aux structures floues et peu visibles, telle des mammographies [87]. Le seuillage local est également utilisé comme étape finale, car il permet un traitement adaptatif du résultat d’une suite de filtres [70]. Toujours dans les méthodes classiques, la croissance de région part d’un pixel de l’image, appelé graine, qui appartient à la structure à segmenter, et recherche à partir de là des pixels d’intensité similaires, en suivant un critère d’homogénéité au sein de la structure à segmenter. Ces techniques ont trouvé leur utilité pour des images cardiaques [151], ou angiographiques [71] ; leur robustesse laisse cependant à désirer, et on leur préfère un usage dans le cadre des Interfaces Homme / Machine et des segmentations semi-automatiques [109]. La méthode dite des lignes de partage des eaux (watershed) [15] est également une méthode de segmentation de région très intéressante, car elle permet d’introduire une forme de détection de contour dans la segmentation de région en utilisant le gradient de l’image à segmenter comme représentation topographique [137]. Très dépendante de l’initialisation, elle convient mal pour les images fortement bruitées : le passage au gradient augmente encore ce bruit et rend le résultat difficilement exploitable. Les lignes de partage des eaux sont cependant très utilisées dans le cadre des segmentations interactives, sur des structures bien contrastées [83] ou dans un système semi-automatique .
Logique floue, ensembles statistiques
La théorie des ensembles flous n’est que la généralisation de la théorie des ensembles présentée par Zadeh en 1965 comme une expression mathématique de l’incertitude [178]. Sans entrer dans les détails, elle consiste à définir une mesure floue par une mesure de probabilité Π appliquée sur une partie Y d’un ensemble X (Π(∅) = 0, Π(X) = 1) , représentable par une distribution de possibilité sur l’ensemble X. Ceci permet de travailler sur un espace de caractéristiques, comme des coefficients d’ondelettes [12], et de faire plus facilement de la fusion de données, puisque toutes les informations sont représentées par des distributions de possibilité. Pour obtenir un résultat final, il faut appliquer aux distributions un algorithme de partitionnement. La solution la plus simple est un algorithme dérivé du fuzzy c-means, qui fournit un partitionnement non-supervisé, puisque la formation des différentes classes n’est pas guidée par un expert. Cet algorithme, célèbre par sa simplicité, est souvent utilisé comme première étape à un processus de segmentation [7], comme base à un algorithme plus complexe [119, 118], ou à titre de comparaison [177]. La littérature est riche quand à l’utilisation des techniques dérivées des ensembles flous appliquées à la segmentation [16, 67, 163]. Une forme de recherche de graphe associée à un système basé sur la logique floue conduit à un système de CHAPITRE 4. SEGMENTATION EN TISSUS 38 segmentation perfectionné [163] qui a mené à une étude sur une très grande base de données appliquée à la sclérose en plaques [162] dans le cadre d’un test clinique, ce qui souligne la fonctionnalité de la méthode. Des critères flous peuvent être facilement introduits dans un processus de segmentation différent, à base de modèles déformables, voir par exemple [33]. Enfin, on ne compte plus le nombre de technique d’extraction des inhomogénéités dans les IRM à base de fuzzy cmeans [1, 86, 144]. D’un point de vue statistique, l’algorithme de fuzzy c-means peut être vu comme un partitionnement flou basé sur l’écart à la moyenne. Dans [68], Hathaway présente l’algorithme d’Espérance Maximisation (EM) comme une minimisation alternée du même critère que celui utilisé dans fuzzy c-means auquel on ajoute une pénalité sous un terme entropique. Désormais, l’estimation des paramètres du modèle (moyenne, matrice de covariance, probabilité a priori) fait partie intégrante de l’algorithme, et les extensions sont multiples. Même si d’autres études l’ont précédé [58, 85], Wells a été le premier à intégrer une correction des inhomogénéités des IRM à partir des bases mathématiques posées par Dempster [39]. De nombreux travaux ont alors suivi, notamment sur l’utilisation des champs de Markov pour ajouter des contraintes spatiales [141, 164], intégrer des modèles de volume partiel [60, 40] ou segmenter des lésions de tout type [103, 43, 167]. De récentes études ont également montré qu’il était possible de coupler un système de segmentation basé sur l’EM avec un algorithme de recalage non-rigide, pour spécifier plus précisément un a priori et intégrer une labélisation plus précise de structures anatomiques [124]. Facile à implémenter et mathématiquement solide, l’EM est une base intéressante pour la segmentation, et en particulier la segmentation de tissus cérébraux.
Modèles déformables
Les modèles déformables introduits par [77], encore appelés snakes, contours actifs ou ballons, ont connu de nombreuses applications en segmentation, mais aussi en animation, suivi de contour ou modélisation géométrique. Ils sont en effet généralement plus robustes au bruit et aux éventuelles discontinuités dans les contours de l’image. Ils permettent en outre une interaction relativement aisée avec l’utilisateur ainsi que l’introduction de connaissances a priori concernant la forme de l’objet recherché. Enfin, on peut obtenir en théorie une segmentation sub-voxellique de l’image. Deux grandes familles de modèles déformables existent : les modèles paramétriques [77, 56] et les les modèles géométriques [91, 24]. Les premiers sont les plus anciens et nécessitent une représentation paramétrique ou discrète. Les seconds, fondés sur la théorie d’évolution des courbes et la méthode des ensembles de niveau (level sets) introduite par Osher et Sethian , utilisent une représentation implicite du modèle et permettent des changements de topologie. Pitiot nous donne une très bonne description des paradigmes associés aux modèles déformables. Cependant, si de récentes études montrent que les modèles déformables et les ensembles de niveau sont parfaitement utilisables dans le cadre de la segmentation de tissus cérébraux, leur mise en œuvre reste difficile et les temps de calcul sont souvent très longs. De plus, la variabilité des tissus cérébraux, notamment des replis du cortex, est très difficile à intégrer dans un modèle de ce type, et est un sujet d’étude à part entière. Quand à la segmentation des lésions de SEP, qui reste notre but final, elle est difficile par ce biais, car il n’existe pas d’a priori suffisamment précis sur la forme des différentes lésions. Il est cependant possible d’introduire ce genre de contrainte dans un système semi-automatique, ou en intégrant la forme des structures environnantes [122], mais le processus est très lourd à mettre en œuvre.
Discussion
Rappelons brièvement les données du problème final, la segmentation des lésions de sclérose en plaques en IRM multi-séquences. Ces lésions sont présentes essentiellement dans la matière blanche, et sont couramment définies dans la littérature médicale comme des hypersignaux de la substance blanche. Mais comme nous le verrons plus en détail dans le chapitre 6, les lésions de SEP n’ont pas une signature fixe : selon leur localisation (à proximité du cortex, près des ventricules, sous la tente du cervelet), selon le type de maladie observé (rémittente, secondaire progressive), le signal des lésions est difficile à prévoir. Il est toujours possible d’utiliser une grande base de données de patients et de témoins dans le cadre d’un système expert ou basé sur des réseaux de neurones. De telles applications ont déjà vu le jour, et leur fonctionnalité a été montrée [179, 181]. De telles méthodes, bien que disponibles, sont cependant restreintes à l’utilisation de grandes bases de données pour les besoins d’apprentissage du système, et requièrent une certaine uniformité dans la qualité des images initiales. Nous avons préféré choisir une solution moins restrictive par rapport aux données utilisées. Des techniques basées sur des modèles déformables existent, mais leur paramétrisation est délicate, les temps de calcul sont très longs et la qualité des images dont nous disposons nous laisse penser que des solutions basées sur l’intensité et travaillant à l’échelle du voxel sont envisageables. L’algorithme d’Espérance Maximisation est souvent pris en exemple pour sa robustesse quand il est contraint par un atlas statistique comme probabilité a priori. La preuve de convergence, la possibilité d’y inclure une correction des inhomogénéités de l’image ainsi que des contraintes spatiales en font un algorithme de choix pour la segmentation des tissus cérébraux. Une faiblesse de l’EM est pourtant la nécessité que l’atlas soit représentatif des patients à segmenter, ce qui n’est pas acquis d’avance. Lors de la segmentation par un EM, par exemple, d’images néonatales comme celles étudiées dans [127], un atlas statistique constitué à partir de patients adultes ne convient pas. Lorsque la variabilité entre les patients est trop importante, d’autres méthodes comme les classificateurs kNN supervisés donnent de meilleurs résultats [35]. Cette méthode consiste à comparer chaque voxel à segmenter à l’ensemble d’une base d’apprentissage, et à sélectionner les k éléments de cette base les plus proches pour obtenir une labélisation finale. Ceci permet d’utiliser facilement une base d’apprentissage labélisée, mais sans phase d’apprentissage, comme pour un réseau de neurones par exemple. Cependant, la sclérose en plaques étant une maladie touchant à large majorité les adultes de 20 à 40 ans, l’espace des images à segmenter s’en trouve fortement restreint, et l’atlas fourni par le MNI convient à notre étude. Dans la suite de ce chapitre, une présentation de l’algorithme EM sera effectuée, ainsi que son application à la segmentation des IRM multi-séquences en tissus cérébraux. Une discussion sera menée pour montrer les faiblesses de l’algorithme qu’il faudra pallier.
1 Introduction |